SARS-CoV-2
| Respekt für Li Wenliang (李文亮) † 7.2.2020 |
Synonyme: SARS-assoziiertes Coronavirus 2, 2019-nCoV (obsolet), 2019-novel Coronavirus (obsolet), Wuhan-Coronavirus
Englisch: severe acute respiratory syndrome coronavirus 2, SARS-CoV-2, 2019 novel coronavirus, Wuhan coronavirus
Definition
SARS-CoV-2, vormals 2019-nCoV, ist ein im Jahr 2019 entdecktes Coronavirus, das zur Linie B der Betacoronaviren gehört. Es handelt sich um ein behülltes (+)ssRNA-Virus, das die Coronavirus-Erkrankung COVID-19 auslöst.
Nomenklatur
Das Virus wurde von der WHO und anderen Organisationen zunächst unter der provisorischen Bezeichnung "2019-nCoV" für "novel Coronavirus" geführt. Weitere Virusbezeichnungen waren "Wuhan-Coronavirus", "WH-Human-1 coronavirus" (WHCV) und "Wuhan seafood market pneumonia virus". Ein Namensvorschlag in Anlehnung an MERS-CoV war "Wuhan respiratory syndrome coronavirus", kurz "WRS-CoV". Keine dieser Bezeichnungen konnte sich durchsetzen.
Am 11.2.2020 gab die WHO dem Virus seinen endgültigen Namen SARS-CoV-2. Die von ihm ausgelöste Erkrankung nennt man offiziell "COVID-19" bzw. "Covid-19" für "Corona virus disease 2019". Die Virusbezeichnung wurde am gleichen Tag von der Coronavirus Study Group auf dem Preprint-Server bioRxiv übernommen. Synonyme Bezeichnungen für COVID-19 sind 2019-nCoV-Infektion, Coronavirus-Pneumonie oder Wuhan-Fieber. Englische Alternativbezeichnungen sind "2019-nCoV acute respiratory disease", "coronavirus disease 2019" und "novel coronavirus pneumonia (NCP)".
Herkunft
Die genaue Herkunft des Virus ist bislang unklar (2023). SARS-CoV-2 zeigt phylogenetisch eine große Ähnlichkeit zu mehreren Coronavirus-Stämmen, die in China aus Fledermäusen der Familie Rhinolophidae (Hufeisennasen) isoliert wurden. Dazu zählen BatCoV RaTG13, RmYN02 und RpYN06, die eine genomische Ähnlichkeit von bis zu 96,1 % aufweisen.[1][2] Damit gilt es als wahrscheinlich, dass das Virus aus dieser Wirtstierpopulation stammt. Allerdings wird kein direkter Übergang, sondern ein noch nicht identifizierter Zwischenwirt angenommen. Als mögliche Kandidaten kommen u.a. Schuppentiere (Pangoline), Schleichkatzen, Marderhunde und Katzen in Frage. Isolate aus dem malayischen Schuppentier zeigen eine 99%ige genomische Ähnlichkeit mit der rezeptorbindenden Domäne des humanpathogenen Virus. Die Übereinstimmung der gesamten RNA liegt jedoch nur bei 90 %.[3] Die Theorie, dass Marderhunde eine mögliche Virusquelle sind, erhielt 2023 durch Genomanalysen von Isolaten des Huanan Seafood Market neuen Auftrieb.[4]
Aufgrund bestimmter genetischer Merkmale, insbesondere der relativen synonymen Codonverwendung (RSCU), wurden anfangs auch Schlangen als primäre Wirtstiere diskutiert. Der Speziesübergang soll durch homologe Rekombination des Spike-Proteins S erfolgt sein.[5]
Die mangelnde Identifikation des Zwischenwirts hat dazu geführt, dass auch die mögliche Freisetzung des Virus durch einen Laborunfall diskutiert wird. Als Hinweis darauf wird gewertet, dass im Institut für Virologie Wuhan Forschungen durchgeführt wurden, die gezielte Gain-of-Function-Mutationen von Coronaviren beinhalteten.[6] Ein konkreter Beleg für diese Virusherkunft steht jedoch aus.
Systematik
- Klassifikation: Viren
- Bereich: Riboviria (RNA-Viren)
- Ordnung: Nidovirales
- Familie: Coronaviridae
- Gattung: Betacoronavirus
- Untergattung: Sarbecovirus
- Art: SARS-CoV-2
- Untergattung: Sarbecovirus
- Gattung: Betacoronavirus
- Familie: Coronaviridae
- Ordnung: Nidovirales
- Bereich: Riboviria (RNA-Viren)
Genetik
Virusgenom
Das Virusgenom von SARS-CoV-2 wurde erstmalig am 2.1.2020 vollständig sequenziert und am 11.1.2020 veröffentlicht. Es umfasst rund 30 kbp und kodiert mehrere Proteine, die als Open-Reading-Frame-Proteine (ORF), Nichtstrukturproteine (NSP) und Strukturproteine (S, E, M, N) gelabelt werden:[7][8][9][10]
- 7.096 Aminosäuren (AS) langes ORF1ab-Polyprotein, bestehend aus
- NSP1: 180 AS; Host translation inhibitor, spaltet die RNA der Wirtszelle
- NSP2: 638 AS; mögliche Interaktion mit Prohibitin (PHB)
- NSP3: 1.945 AS; Papain-ähnliche Protease (PL-PRO)
- NSP4: 500 AS; unterstützt wahrscheinlich die Vesikelbildung
- NSP5: 306 AS; 3C-ähnliche Proteinase (3CL-PRO), spaltet den C-Terminus des Polyproteins
- NSP6: 290 AS; beeinflusst die Bildung von Autophagosomen
- NSP7: 83 AS; formt ein Hexadekamer mit NSP8 (8:8), mögliche Primaseaktivität
- NSP8: 198 AS; siehe oben
- NSP9: 113 AS; ssRNA-bindendes Protein
- NSP10: 139 AS; stimuliert die Exoribonuklease (NSP14) und die O-Ribose-Methyltransferase (NSP16)
- NSP12: 932 AS; RNA-abhängige RNA-Polymerase (RdRp)
- NSP13: 601 AS; Helikase, Multifunktionsenzym mit Zinkfingerdomäne
- NSP14: 527 AS; Guanin-N7-Methyltransferase, hat Exoribonuklease-Aktivität
- NSP15: 346 AS; Uridylat-spezifische Endoribonuklease (EndoRNAse)
- NSP16: 298 AS; O-Ribose-Methyltransferase, wichtig für die Immunevasion
- 1.273 AS langes Oberflächen-Glykoprotein (S, "spike")
- 275 AS langes ORF3a-Protein
- 75 langes AS Struktur- bzw. Hüllprotein (E, "envelope")
- 222 AS langes Membran-Glykoprotein (M, "matrix")
- 61 AS langes ORF6-Protein
- 121 AS langes ORF7a-Protein
- 43 AS langes ORF7b-Protein
- 121 AS langes ORF8-Protein
- 419 AS langes Nukleokapsid-Phosphoprotein (N, "nucleoprotein")
- 38 AS langes ORF10-Protein
Am 5'- bzw. 3'-Ende des Genoms befinden sich jeweils kurze untranslatierte Bereiche (UTRs).
Mutationen
Die genetische Varianz von SARS-CoV-2 ist geringer als bei anderen RNA-Viren, da Coronaviren wegen ihres größeren und komplexen Genoms sehr wahrscheinlich über eine Transkriptionsfehlerkorrektur verfügen.[11] Im Rahmen der Virusmutation kommt es vor allem zu Einzelnukleotid-Polymorphismen (SNPs), die nicht-synonym oder synonym sein können, d.h. einen Aminosäurewechsel nach sich ziehen oder nicht.
Durch die Mutationen lässt sich die globale Ausbreitung des Virus verfolgen. Ferner kann man auf ihrer Grundlage verschiedene Virustypen differenzieren. Ob die bislang beobachteten Unterschiede im Virusgenom eine derartige Unterteilung rechtfertigen, ist unter Virologen jedoch umstritten.[12]
Auf Basis der Analyse von mehr als 100 Virusisolaten wurden anfangs zwei genetische Hauptvarianten von SARS-CoV-2 differenziert, die man als L-Typ und S-Typ bezeichnet. Sie unterscheiden sich durch 2 SNPs. Der L-Typ hat sich wahrscheinlich aus dem S-Typ entwickelt und ist die verbreitetere und "aggressivere" Variante.[13]
Eine weitere Studie mit 160 Virusisolaten aus verschiedenen Ländern differenzierte das Virus in einen A-, B- und C-Typ, wobei der A-Typ die älteste Virusvariante darstellt. Der A- und C-Typ waren in Europa und Amerika am verbreitetsten, der B-Typ in Asien.[14]
Das ursprüngliche Nomenklatursystem der verschiedenen Virusvarianten baut auf ihren genetischen Verwandtschaftsbeziehungen auf. Dabei werden die Varianten nach bestimmten Regeln hierarchisch als B.1, B.2, B.3 usw. bezeichnet. Abkömmlinge von B.1 sind dann entsprechend B.1.1, B.1.2 usw. Ab der vierten Hierarchieebene wird ein neue Hauptlinie eröffnet: B.1.1.1.1 würde zum Beispiel zu C.2.[15] Davon unabhängig werden Virusvarianten jedoch auch häufig nach einzelnen Genmutationen benannt.
Es sind mehrere tausend Mutationen bekannt, deren Anzahl stetig weiter ansteigt. Beispiele sind:
- Punktmutationen: L452R, Y453F, E484K, E484Q, Q677P, D796H, T8782C, G26144T oder C28144T
- Deletionen: H69/V70
Die meisten umlaufenden Viren weisen mehrere Mutationen auf. Bestimmte Mutanten sind mit regionalen Ausbrüchen verbunden, z.B. Cluster 5 in Dänemark, B.1.1.7 (VUI-202012/01) im Südosten Englands, 501.V2 in Südafrika oder B.1.617 in Indien.
Die Mutationen ziehen in den meisten Fällen keine signifikante Änderung der Viruseigenschaften nach sich, können aber die Erkennbarkeit durch Laborverfahren reduzieren. Einige Mutationen, die das Spikeprotein betreffen, z.B. D614G oder N501Y führen zu einer erhöhten Infektiosität.[16] Diese Mutanten breiten sich durch Selektion stärker aus als weniger infektiöse Virusstämme. Bei der B.1.1.7-Mutante konnte zudem eine um etwa 60 % gesteigerte Mortalität gegenüber älteren Virusvarianten nachgewiesen werden.[17]
Welchen Einfluss die Mutationen auf die Wirksamkeit der verschiedenen Impfstoffe haben, kann derzeit (2023) noch nicht verlässlich eingeschätzt werden. Impfstoffe erzeugen eine Immunantwort auf mehrere Antigenstrukturen, so dass meist eine höhere Zahl an Mutationen notwendig ist, um ihre Wirkung abzuschwächen.
Besorgniserregende Varianten
Die WHO hat mehrere Virusvarianten, die aus epidemiologischer Sicht bedenklich sind, als besorgniserregende Varianten bzw. "Variants of Concern" (VOC) eingestuft.[18] Kriterien für die Einordnung als VOC sind u.a. die schnellere Verbreitung, die Schwere des klinischen Verlaufs und die Effektivität von Nachweismethoden, Gegenmaßnahmen oder Impfstoffen. Die Nomenklatur orientiert sich am griechischen Alphabet:[19]
- Alpha-Variante (B.1.1.7, 501Y.V1, VUI-202012/01) - erstmals Mitte Dezember 2020 in Großbritannien nachgewiesen
- Beta-Variante (B.1.351, 501Y.V2) - erstmals im Dezember 2020 in Südafrika nachgewiesen
- Gamma-Variante (P1, 501Y.V3) - erstmals in Brasilien nachgewiesen
- Delta-Variante (B.1.617.2) - erstmals in Indien nachgewiesen
- Omikron-Variante (B.1.1.529) - erstmals im November 2021 in Botswana und Südafrika aufgetreten
Die VOCs unterscheiden sich in ihren Eigenschaften teilweise deutlich vom Ursprungsvirus. Unter anderem fallen sie durch eine erhöhte Übertragbarkeit, eine stärkere Virulenz und eine verringerte Effektivität neutralisierender Antikörper auf. Dadurch können sie einer veränderten Krankheitsschwere (Fallsterblichkeit und Hospitalisierungsrate) führen. Wie diese Veränderung sich klinisch darstellt, zeigt sich in der Regel erst nach der Ausbreitung und einer eingehenden Analyse des mutierten Virus. So weist z.B. die Omikron-Variante eine höhere Übertragbarkeit im Vergleich zur zuvor dominierenden Delta-Variante auf, führt jedoch in den meisten Fällen zu einem etwas milderen Verlauf.
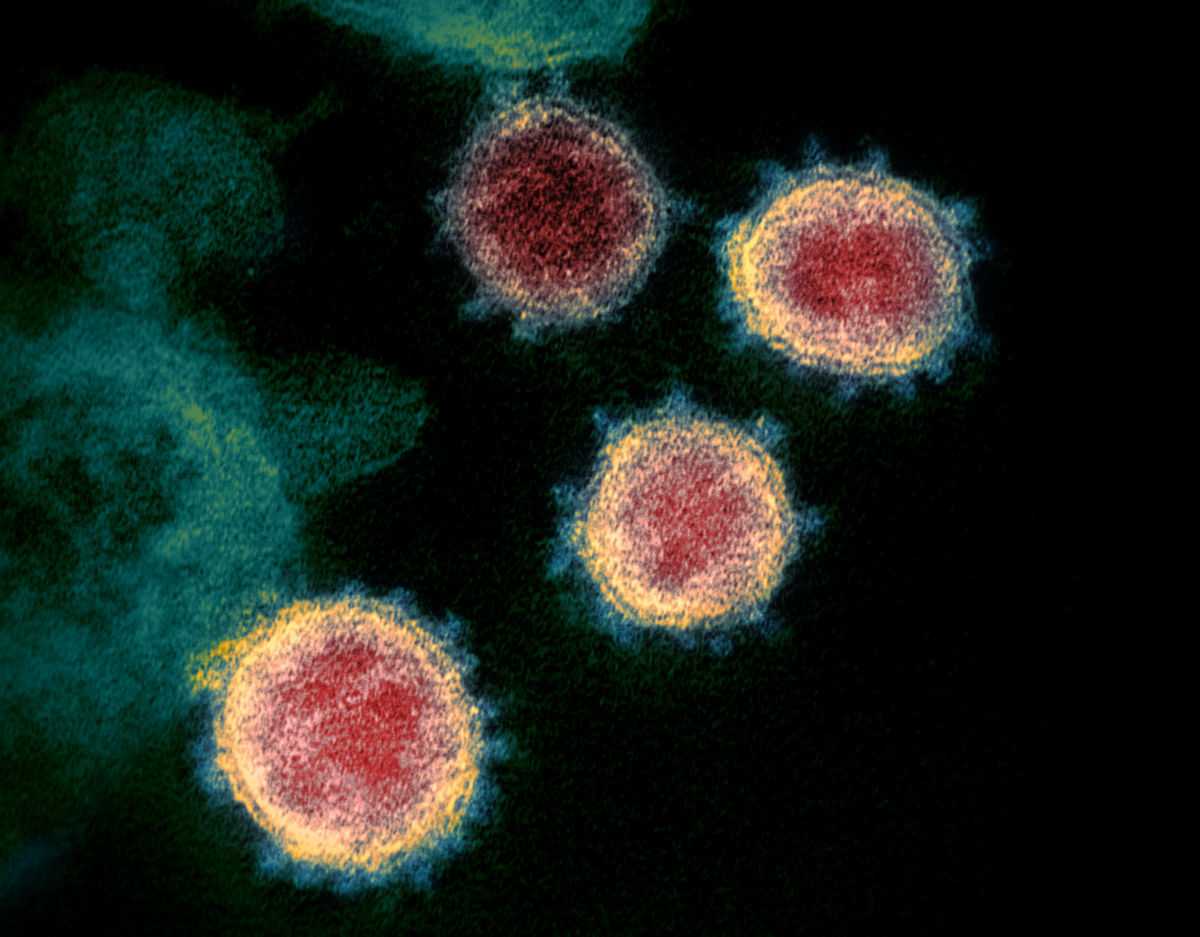
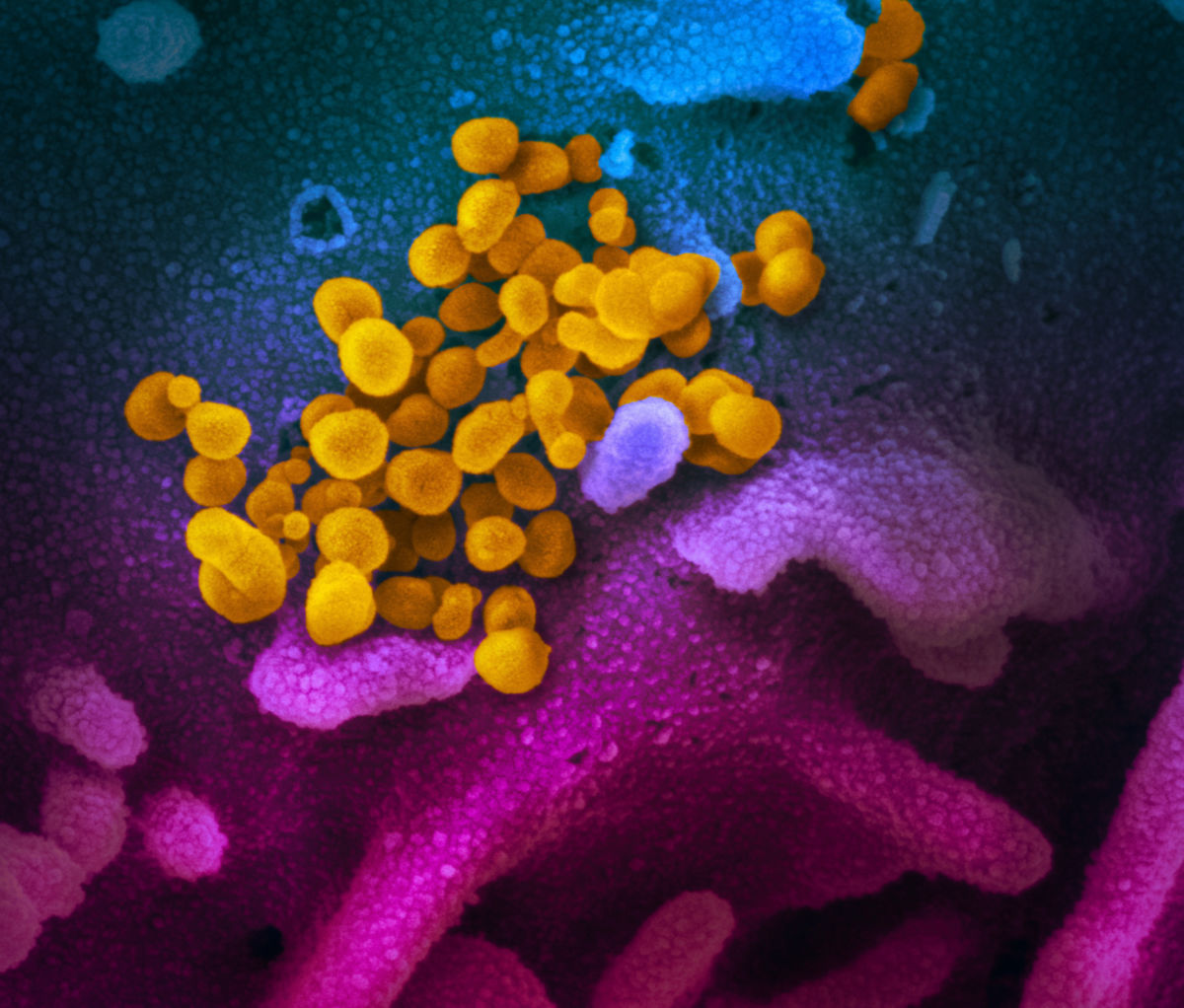
Morphologie
In der Elektronenmikroskopie stellen sich die Viruspartikel als rundliche Strukturen mit einem Durchmesser von 60 bis 140 nm dar. Diese Größenunterschiede sind wahrscheinlich darauf zurückzuführen, dass bei der Knospung (Budding) nicht alle Partikel ein Nukleokapsid enthalten.[20] Es zeigt sich eine leichte Pleomorphie. Die Virushülle besteht aus einer Lipiddoppelschicht, aus der die Spikes des Virus etwa 9 bis 12 nm hervorragen.[21] Es finden sich im Durchschnitt nur insgesamt 26 solcher Spikes auf der Oberfläche, dazwischen liegen größere Lücken.[22] Die Spikeproteine bestehen aus mehreren Untereinheiten:
- einem kurzen Verankerungselement,
- einer S1-Einheit, welche die rezeptorbindende Domäne (RBD) trägt und
- einer homotrimeren S2-Einheit aus Glykoproteinen, welche die Fusion mit der Wirtszellmembran ermöglicht.
Die Spikeproteine sind sehr flexibel, der Kopf kann sich in alle Richtungen bewegen. Zudem ragen die Spikes nicht senkrecht aus der Membran heraus, sondern sind durchschnittlich um 40° zur Seite geneigt.[22]
Ferner finden sich in der Virushülle weitere Proteinbestandteile wie M-Proteine, E-Proteine und Hämagglutininesterase-Dimere (HE).
Die spiralig aufgewundene RNA im Virusinneren ist von einem Nukleokapsid aus N-Proteinen umgeben.
Kultivierung
Das Virus kann zu Forschungszwecken in einer Zellkultur vermehrt werden.[23] Für die Anzucht lassen sich Epithelzellen der menschlichen Atemwege sowie Vero- und Huh7-Zellen einsetzen.[24]
Übertragung
Der Hauptübertragungsweg von SARS-CoV-2 ist die Weitergabe von Mensch-zu-Mensch. Eine Übertragung von Tier-zu-Mensch sowie die Übertragung zwischen Tieren ist ebenfalls möglich.
...durch Menschen
Die Erreger können zwischen Menschen auf folgende Weise weitergegeben werden:
- Tröpfcheninfektion: Einatmen infizierter Flüssigkeitspartikel (> 5 µm Durchmesser), die ein Erkrankter durch Niesen oder Husten verbreitet. Dabei handelt es sich wahrscheinlich um den Hauptübertragungsweg. Die Viruskonzentration in den Atemwegen der Infizierten erreicht etwa 5 bis 6 Tage nach Einsetzen der Symptome ihr Maximum.[25][26] Der kritische Abstand zum Erkrankten wird mit 1,8 m angegeben.[27] Infektiöse Sekrete können auch bei endotrachealer Intubation, nicht-invasiver Beatmung oder Absaugen aus den Atemwegen entstehen.
- Aerogene Infektion: Eine Übertragung über zirkulierende Luft ist sehr wahrscheinlich. Hinweise darauf sind, dass sich die Infektion sehr schnell in geschlossenen Räumen mit ungefiltertem zirkulierendem Luftstrom (Kreuzfahrtschiffe, Fleischereibetriebe, Gaststätten) verbreitet. In dokumentierten Einzelfällen wurden Viren in einem Radius von mehr als 8 Metern übertragen.[28] SARS-CoV-2 ist wie SARS-CoV-1 in Aerosolen (Partikel < 5 µm Durchmesser) über mindestens 3 Stunden nach der Entstehung des Aerosols nachweisbar.[29][30] Die Viruskonzentration im Aerosol lag dabei auch am Ende des Beobachtungszeitraums noch über der minimalen Infektionsdosis in der Zellkultur (TCID50). Infektiöse Viruspartikel ließen sich auch in der Luft normaler Krankenhauszimmer nachweisen, in denen COVID-19-Patienten untergebracht waren.[31] Bei allen Prozessen mit Aerosolbildung (z.B. Bronchoskopie, zahnmedizinische Behandlung) wird deshalb das Tragen einer Atemschutzmaske und Schutzbrille empfohlen.[32][33]
- Kontaktinfektion: Enger körperlicher Kontakt bzw. Berührung eines Erkrankten, anschließende Autoinokulation durch Verschleppung des Erregers auf die Schleimhaut.
- Schmierinfektion: Berührung kontaminierter Gegenstände. Coronaviren können 2 bis 3 Tage, nach anderen Studien sogar bis zu 9 Tage auf Oberflächen wie Metall, Glas oder Plastik überleben.[34] Auf Pappe beträgt die Nachweisdauer bis zu 24 Stunden, auf Kupferoberflächen bis zu 4 Stunden.[29] Die Viruslast auf allen Oberflächen nimmt dabei mit zunehmender Zeit ab.
Ein fäkal-oraler Übertragungsweg kommt ebenfalls in Frage, da sich SARS-CoV-2 im Stuhl von Infizierten nachweisen lässt.[35] Dieser Infektionsweg spielt aber wahrscheinlich, wie bei SARS, für die Verbreitung von COVID-19 eine untergeordnete Rolle.
Darüber hinaus wurde mehrfach eine mögliche vertikale Virusübertragung von der Mutter auf das ungeborene Kind beschrieben.[36][37] Die Übertragung scheint aber nicht der Regelfall zu sein - eine Studie mit 9 Schwangeren fand keinen Hinweis auf eine vertikale Übertragung.[38]
...durch Tiere
Eine Übertragung von COVID-19 durch Haustiere auf den Menschen oder umgekehrt ist grundsätzlich möglich. ACE2, das Zielprotein von SARS-CoV-2, hat bei Menschen und Katzen eine hohe Strukturhomologie.[39] Tierexperimentelle Studien ergaben, dass Katzen und Frettchen an COVID-19 erkranken können. Die hohe Empfänglichkeit der Unterfamilie Mustelinae für SARS-CoV-2 zeigte sich durch die rasche Durchseuchung europäischer Nerzfarmen. Eine signifikante Virusreplikation bei Hunden, Schweinen und Geflügel ist hingegen nicht nachweisbar.[40]
SARS-CoV-2 vermehrt sich im Respirationstrakt von Frettchen und Katzen und führt dort zu einer relevanten Viruslast. Es wird unter Nerzen und Frettchen durch Tröpfcheninfektion weitergegeben, wahrscheinlich auch unter Katzen.[40] Ferner lässt es sich im Kot der erkrankten Tiere nachweisen. Ein Übergang des Virus auf den Menschen ist bei Nerzen belegt.[41] Für andere Tierarten kann er nicht ausgeschlossen werden. Daher sollte man – vor allem beim Vorliegen von Risikofaktoren – einen engen Kontakt mit potentiell infizierten Tieren vermeiden.
Kontagiosität
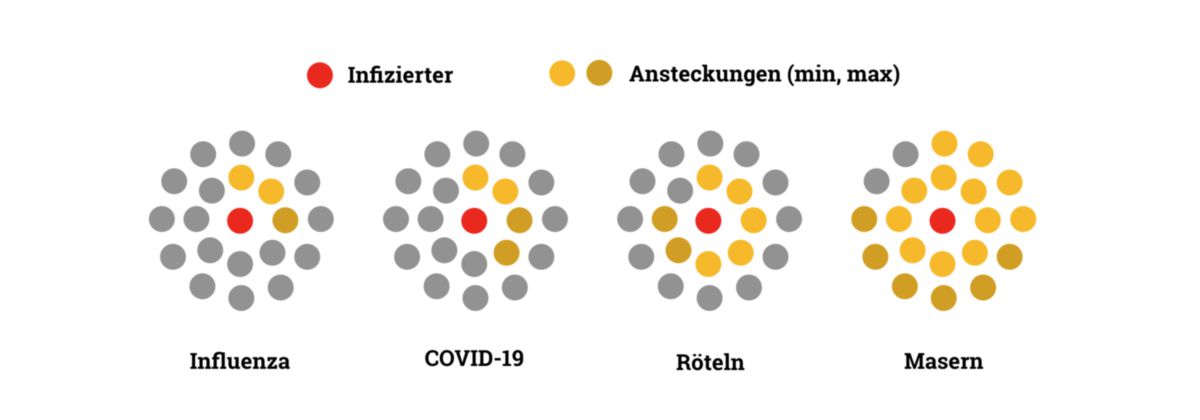
SARS-CoV-2 hat eine vergleichbare Übertragbarkeit von Mensch zu Mensch wie das SARS-Coronavirus (SARS-CoV). Die Basisreproduktionszahl (R0) des Virus wurde zunächst zwischen 3 und 5, später zwischen 1,5 und 3,5 angegeben.[42][43] Die WHO nennt eine etwas niedrigere Range von 1,4 bis 2,5.[44] Ein Infizierter gibt die Erkrankung wahrscheinlich im Durchschnitt an etwa 2 bis 4 Nicht-Infizierte weiter. Wie bei SARS sind Superspreader jenseits der Basisreproduktionszahl möglich, was das plötzliche Auftreten größerer lokaler Epidemieherde erklärt.
Infektiosität
Zur Infektiosität von SARS-CoV-2 gibt es zur Zeit (2023) keine verlässlichen Zahlenangaben. Eine konkrete Festlegung wird dadurch erschwert, dass sich die verschiedenen zirkulierenden Virusvarianten in ihrer Infektiosität unterscheiden. Die minimale Infektionsdosis, d.h. die Zahl der Viruspartikel, die eine Infektion auslösen, wird zwischen einigen hundert oder tausend Viren geschätzt.[45] Die initial übertragene Virusdosis hat wahrscheinlich einen wichtigen Einfluss auf den Krankheitsverlauf. Eine kleinere Virusdosis lässt dem Immunsystem eine längere Reaktionszeit und korreliert mit leichteren Verläufen, während eine größere Virusdosis potentiell schwere Verläufe auslöst.[46]
Die Burst Size von SARS-CoV-2 ist nicht bekannt.
Infektionsablauf
Für das Andocken des Virus an menschliche Epithelzellen ist das Spike-Protein S des Virus verantwortlich. Es bindet an die Exopeptidase ACE2 menschlicher Zellen. Strukturanalysen zeigen, dass die Affinität höher ist im Vergleich zum SARS-Coronavirus, das ebenfalls an dieses Protein bindet.[47] Im Rahmen der Rezeptorbindung unterliegt das Spikeprotein mehreren Konformationsänderungen, die man als S1, S2 und S2' bezeichnet.[10] Zwischen der S1- und S2-Untereinheit gibt es einen polybasischen Spaltbereich (Furin-Spaltstelle), der spezifisch für SARS-CoV-2 ist.[48]
Der Eintritt in die Wirtszelle wird durch proteolytische Spaltung des Spikeproteins an der S1/S2- und S2'-Stelle vermittelt. Damit diese Spaltung im Rahmen der Infektion erfolgen kann, muss die Aktivierungssequenz vorher durch Furin aufgebrochen werden.[49] Durch Konformationsänderung der S1-Domäne wird daraufhin die rezeptorbindende Domäne freigesetzt und interagiert mit ACE2. Mithilfe der Serinprotease TMPRSS2 wird daraufhin die zweite Spaltung ermöglicht, möglicherweise sind an diesem Schritt ebenfalls Furin-ähnliche Enzyme beteiligt. Dies führt wiederum zur Konformationsänderung der S2-Domäne, die nachfolgend die Fusion mit der Wirtszellmembran vermittelt. Furin und TMPRSS2 wirken synergistisch und der kombinierte Einsatz von Inhibitoren für beide Enzyme konnte die SARS-CoV-2 Infektion in Zellkultur effizient unterbinden.[50] Die genauen Mechanismen, die zur Fusion der Virushülle mit der Zellmembran der Wirtszelle führen, sind jedoch noch nicht vollständig geklärt. Nach der erfolgreichen Fusion wird das Nukleokapsid des Virus in das Zytoplasma freigesetzt.
Im Zytoplasma wird die Produktion viraler Proteine durch die Translationsmachinerie der Wirtszelle eingeleitet. Die gebildeten Replikasen wandern zum endoplasmatischen Retikulum (ER), wo sie "Replikationsfabriken" bilden. Diese enthalten Doppelmembranvesikel, in denen die Transkription stattfindet. Dabei entsteht doppelsträngige RNA (dsRNA), die in den Vesikeln vor der Erkennung durch Pattern-Recognition-Rezeptoren abgeschirmt sind. Kommt es zur Erkennung durch das Immunsystem, wird eine Signalkaskade in Gang gesetzt, die zur Produktion verschiedener Interferone und Chemokine führt.
Die viralen Oberflächenproteine werden am ER in die Membran eingefügt. Daraufhin findet am ER-Golgi-Zwischenkompartiment (ERGIC) der Zusammenbau des Viruspartikels statt ("virion assembly"). Die Viruspartikel werden durch Exozytose wieder aus der Zelle herausgeschleust.
Bei Patienten, die Arzneistoffe einnehmen, welche die Expression von ACE2 verstärken, zum Beispiel ACE-Hemmer oder Sartane, wurde ein höheres Infektionsrisiko postuliert.[51] Verschiedene Analysen mehrerer tausend Erkrankungsfälle liefern jedoch keinen Hinweis darauf, dass das Erkrankungsrisiko oder die Schwere des Krankheitsverlaufs durch ACE-Hemmer beeinflusst werden.[52][53][54] Die betreffenden Fachgesellschaften sehen entsprechend keinen Handlungsbedarf, eine bestehende antihypertensive Medikation zu modifizieren.[55][56]
Gewebetropismus
Das Virus adressiert in erster Linie die Nasenschleimhaut, die Rachenschleimhaut und das respiratorische Epithel der unteren Atemwege. Es scheint dabei ein stärkerer Tropismus für die oberen Atemwege vorzuliegen als bei SARS-CoV, der an Influenzaviren erinnert.[57] Dadurch ist die Virusausscheidung wahrscheinlich höher als bei anderen Coronaviren. Das Riechepithel ist durch seine hohe Dichte an ACE2-Rezeptoren ebenfalls bevorzugt betroffen.[58] Weitere Zielzellen des Virus sind die Enterozyten der Dünndarmschleimhaut, vor allem im proximalen und distalen Dünndarm.[59] Mit seiner weiteren Verbreitung im Körper kann das Virus darüber hinaus Perizyten im Kapillarbett verschiedener Organe befallen.[60]
Inkubationszeit
Die Inkubationszeit von der Infektion bis zum Eintreten der ersten Symptome beträgt im Mittel etwa 3 bis 5 Tage.[61][62] Bei schwerem Verlauf erfolgt die Hospitalisation dann meist nach weiteren 4 bis 5 Tagen.[63] Die Inkubationszeit unterliegt jedoch großen interindividuellen Schwankungen, so dass ein Zeitraum von 2 bis 14 Tagen möglich ist.[64] In sehr seltenen Fällen soll die Inkubationszeit bis zu 24 Tagen betragen.[61]
Die Viren können sehr wahrscheinlich bereits während der Inkubationszeit, d.h. vor Ausbruch der Symptome, weitergegeben werden.[65] Diese Annahme wird durch den Nachweis von Viren in Nasenabstrichen asymptomatischer oder minimal symptomatischer Patienten unterstützt.[57][66]
Klinik
Die Ausprägung der Erkrankung ist interindividuell sehr unterschiedlich. COVID-19 kann mit minimalen Symptomen, moderat oder schwer verlaufen. Auch asymptomatische Fälle sind möglich.[67]
Der individuelle Verlauf der Erkrankung ist u.a. von der Viruslast (Inokulum), der Empfänglichkeit des Wirts, der Eintrittspforte der Infektion, der Virulenz des Erregers sowie vom Immunstatus des Wirts abhängig. Zudem verursachen die verschiedenen Virusmutanten unterschiedliche Verläufe, so dass sich das typische Symptombild im Lauf der Pandemie graduell verändert. Im direkten Vergleich zwischen Delta- und Omikron-Variante zeichnet sich letztere dadurch aus, dass mehr asymptomatische Fälle auftraten und die vorliegenden Symptome weniger schwerwiegend und von kürzerer Dauer waren.[68]
Neben Allgemeinsymptomen wie Fieber, Fatigue, Myalgien, Arthralgien, Kopfschmerzen und Schüttelfrost kommt es vor allem zu pulmonalen (Husten, Auswurf, Dyspnoe) und gastrointestinalen Symptomen (Nausea, Diarrhö, Erbrechen, Inappetenz) .
Zudem geht COVID-19 mit einer Hyperkoagulabilität einher, die wahrscheinlich durch die systemische Entzündungsreaktion und eine endotheliale Dysfunktion verursacht wird. Post mortem ließen sich bei etwa 60 % der erkrankten Patienten tiefe Beinvenenthrombosen feststellen. Ferner waren Lungenembolien sowie Mikrothromben in den kleinen Lungenarterien nachweisbar.[69]
siehe Hauptartikel: COVID-19
Virusausscheidung
Die Dauer der Virusausscheidung (Shedding) nach Beginn der Symptome beträgt bei milden Krankheitsverläufen etwa 7 bis 12 Tage, in schweren Fällen bis zu 14 Tagen. Im Einzelfall ist eine prolongierte Virusausscheidung möglich. Bei Kindern kann die Dauer der Virusausscheidung bei milden Verläufen mehr als 4 Wochen betragen.[70]
Die Viruslast symptomatischer und asymptomatischer Patienten unterscheidet sich nicht signifikant.[71]
Risikogruppe
SARS-CoV-2 wird in Deutschland in die Risikogruppe 3 der Biostoffverordnung (BioStoffV) eingeordnet, da eine leichte Übertragbarkeit sowie begrenzte Therapiemöglichkeiten bestehen. Die Arbeit mit lebenden Viren in Zellkulturen verlangt daher ein Sicherheitslabor, das die Schutzstufe 3 (S3-Labor) ermöglicht.
Für den Umgang mit infektiösem Untersuchungsmaterial in der Labordiagnostik (z.B. Probenaufbereitung für die RT-PCR) ist die Schutzstufe 2 ausreichend. Arbeitsabläufe, bei denen eine Aerosolbildung möglich ist, müssen an einer mikrobiologischen Sicherheitswerkbank der Klasse II erfolgen. Dabei ist eine persönliche Schutzausrüstung zu tragen.
Literatur
Bildquellen
- NIAID, Creative Commons CC 2.0
- 3D-Illustration SARS-CoV-2, DocCheck CC BY-NC-ND
- NIAID Rocky Mountain Laboratories (RML), U.S. NIH; Public Domain
- Animation eines Virusinfekts, DocCheck CC BY-NC-ND
Quellen
- ↑ Roujian Lu et al. Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding, The Lancet 2020
- ↑ Peng Zhou et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin Nature 2020
- ↑ David Cyranosk, Mystery deepens over animal source of coronavirus, Nature News, abgerufen am 6.3.2020
- ↑ Smriti Mallapaty: COVID-origins study links raccoon dogs to Wuhan market: what scientists think. Nature News, 21.3.2023, abgerufen am 29.3.2023
- ↑ Wei Ji et al. Cross‐species transmission of the newly identified coronavirus 2019‐nCoV, Journal of Medical Virology 2020
- ↑ David Cyranoski The biggest mystery: What it will take to trace the coronavirus source. Nature News 2020
- ↑ Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome; GenBank: MN908947.3
- ↑ Novel coronavirus complete genome from the Wuhan outbreak now available in GenBank. NCBI Insights, posted on 13. Januar 2020, abgerufen am 21. Januar 2020
- ↑ NCBI: 2019-nCoV Sequences (Wuhan coronavirus), abgerufen am 8.2.2020
- ↑ 10,0 10,1 Swiss-Model: Severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2), abgerufen am 27.2.2020
- ↑ Everett Clinton Smith et al. Coronaviruses Lacking Exoribonuclease Activity Are Susceptible to Lethal Mutagenesis: Evidence for Proofreading and Potential Therapeutics PLOS Pathogens 2013
- ↑ Oscar A. MacLean et al.: Response to "On the origin and continuing evolution of SARS-CoV-2", virological.org, abgerufen am 11.4.2020
- ↑ Tang X et al.: On the origin and continuing evolution of SARS-CoV-2 National Science Review 2020
- ↑ Foster P et al.: Phylogenetic network analysis of SARS-CoV-2 genomes. PNAS 2020
- ↑ Rambaut, A. et al. A dynamic nomenclature proposal for SARS-CoV-2 lineages to assist genomic epidemiology. Nat Microbiol 2020
- ↑ Zhang L et al.: The D614G mutation in the SARS-CoV-2 spike protein reduces S1 shedding and increases infectivity. Scripps Research, abgerufen am 16.6.2020
- ↑ Challen R et al: Risk of mortality in patients infected with SARS-CoV-2 variant of concern 202012/1: matched cohort study BMJ 2021
- ↑ WHO SARS-CoV2 Variants, abgerufen am 11.11.2021
- ↑ RKI: Virologische Basisdaten sowie Virusvarianten abgerufen am 11.11.2021
- ↑ Cornelis A. M. de Haan et al. Assembly of the Coronavirus Envelope: Homotypic Interactions between the M Proteins, J Virol. 2000
- ↑ Na Zhu et al. A Novel Coronavirus from Patients with Pneumonia in China NEJM January 2020
- ↑ 22,0 22,1 Coronavirus Structural Task Force - Das Coronavirus als 3D-Modell 2.0, abgerufen am 25.08.2022
- ↑ Jef Akst: Australian Lab Cultures New Coronavirus as Infections Climb, The-Scientist, 29.1.2020, abgerufen am 1.2.2020
- ↑ EurekAlert: Institut Pasteur isolates strains of coronavirus 2019-nCoV detected in France, 31.1.2020, abgerufen am 12.2.2020
- ↑ Pan Y et al. Viral load of SARS-CoV-2 in clinical samples, The Lancet 2020
- ↑ Kai-Wang K et al. Temporal profiles of viral load in posterior oropharyngeal saliva samples and serum antibody responses during infection by SARS-CoV-2: an observational cohort study, The Lancet 2020
- ↑ Centers for Disease Control and Prevention: Transmission of Novel Coronavirus (2019-nCoV)", 27 January 2020, abgerufen am 22.2.2020
- ↑ Guenther T et al.: Investigation of a superspreading event preceding the largest meat processing plant-related SARS-Coronavirus 2 outbreak in Germany. SSRN Elsevier, posted 23.7.2020, abgerufen am 27.7.2020
- ↑ 29,0 29,1 van Doremalen N et al. Aerosol and Surface Stability of SARS-CoV-2 as Compared with SARS-CoV-1 N Engl J Med 2020
- ↑ Li X. Huang et al.: Role of air distribution in SARS transmission during the largest nosocomial outbreak in Hong Kong, Indoor AirVolume 2004
- ↑ Lednicky JA et al.: Viable SARS-CoV-2 in the air of a hospital room with COVID-19 patients. Int J Infect Dis 2020
- ↑ Way RS et al. Practical recommendations for critical care and anesthesiology teams caring for novel coronavirus (2019-nCoV) patients Can J Anesth 2020
- ↑ Peng X et al. Transmission routes of 2019-nCoV and controls in dental practice, Int J Oral Sci 2020
- ↑ Kampf G, Steinmann E. Persistence of coronaviruses on inanimate surfaces and its inactivation with biocidal agents, Journal of Hospital Infection 2020
- ↑ Gale J: Coronavirus Lurking in Feces May Reveal Hidden Risk of Spread; Bloomberg News February 1, 2020, abgerufen am 2.2.2020
- ↑ Kashmira Gander: Fears Coronavirus could be contracted in the womb after day-old baby in Wuhan tests positive. Newsweek Health, 5.2.2020, abgerufen am 6.2.2020
- ↑ Kirtsman M et al.: Probable congenital SARS-CoV-2 infection in a neonate born to a woman with active SARS-CoV-2 infection CMAJ 2020
- ↑ Huijun Chen et al.: Clinical characteristics and intrauterine vertical transmission potential of COVID-19 infection in nine pregnant women: a retrospective review of medical records The Lancet 2020
- ↑ Andersen KG et al.: The proximal origin of SARS-CoV-2 Nature Medicine (2020)
- ↑ 40,0 40,1 Jianzhong Shi et al.: Susceptibility of ferrets, cats, dogs, and other domesticated animals to SARS–coronavirus 2 Science 2020
- ↑ Detection of new SARS-CoV-2 variants related to mink. ECDC 12 November 2020, abgerufen am 16.12.2020
- ↑ Shi Zhao et al. Preliminary estimation of the basic reproduction number of novel coronavirus (2019-nCoV) in China, from 2019 to 2020: A data-driven analysis in the early phase of the outbreak Int J Infect Dis 2020
- ↑ Imperial College London, Report 3: Transmissibility of 2019-nCoV, abgerufen am 26.1.2020
- ↑ WHO Statement on the meeting of the International Health Regulations (2005) Emergency Committee regarding the outbreak of novel coronavirus (2019-nCoV), 23.1.2020, abgerufen am 10.2.2020
- ↑ van Schaik W in "Expert reaction to questions about covid-19 and viral load; Science Media Centre, 24.3.2020
- ↑ Joshua D. Rabinowitz, Caroline R. Bartman: These Coronavirus Exposures Might Be the Most Dangerous. New York Times 1.4.2020, abgerufen am 5.4.2020
- ↑ Wrapp D et al. Cryo-EM Structure of the 2019-nCoV Spike in the Prefusion Conformation Science 2020
- ↑ Andersen KG et al. The proximal origin of SARS-CoV-2, Nature Medicine 2020
- ↑ Diederich S.: Aktivierung des SARS-Coronavirus 2 aufgeklärt. Leibniz-Institut für Primatenforschung, abgerufen am 8.5.2020
- ↑ Essalmani et al. Distinctive Roles of Furin and TMPRSS2 in SARS-CoV-2 Infectivity J Virol 2022
- ↑ Fang, L. et al. Are patients with hypertension and diabetes mellitus at increased risk for COVID-19 infection?, The Lancet 2020
- ↑ Mehra MR et al.: Cardiovascular Disease, Drug Therapy, and Mortality in Covid-19; NEJM 2020
- ↑ Mancia G et al.:Renin–Angiotensin–Aldosterone System Blockers and the Risk of Covid-19; NEJM 2020
- ↑ BRACE-CORONA-Studie, abgerufen am 14.09.2020
- ↑ ESC Position Statement of the ESC Council on Hypertension on ACE-Inhibitors and Angiotensin Receptor Blockers, abgerufen am 27.03.2020
- ↑ The Renal Association, UK position statement for patients: novel corona virus infection and the use of blood pressure medications. abgerufen am 27.03.2020
- ↑ 57,0 57,1 Lirong Zou et al. SARS-CoV-2 Viral Load in Upper Respiratory Specimens of Infected Patients, N Engl J Med 2020
- ↑ Mengfei Chen et al.:Elevated ACE2 expression in the olfactory neuroepithelium: implications for anosmia and upper respiratory SARS-CoV-2 entry and replication European Respiratory Journal 2020
- ↑ Liang W et al. Diarrhea may be underestimated: a missing link in 2019 novel coronavirus Gut 2020
- ↑ He L et al. Pericyte-specific vascular expression of SARS-CoV-2 receptor ACE2 – implications for microvascular inflammation and hypercoagulopathy in COVID-19 patients bioRxiv 2020, abgerufen am 31.05.2022
- ↑ 61,0 61,1 Wei-jie Guan et al. Clinical characteristics of 2019 novel coronavirus infection in China, NEJM 2020
- ↑ Lauer SA et al. The Incubation Period of Coronavirus Disease 2019 (COVID-19) From Publicly Reported Confirmed Cases: Estimation and Application, Annals of Internal Medicine 2020
- ↑ Natsuko Imai et al. Estimating the potential total number of novel Coronavirus (2019-nCoV) cases in Wuhan City, China Imperial College London, abgerufen am 31.05.2022
- ↑ European Center for Disease Prevention and Control: Rapid Risk Assessment. Outbreak of acute respiratory syndrome associated with a novel coronavirus, Wuhan, China; first update, 22 January 2020, abgerufen am 27.1.2020
- ↑ South China Morning Post: Coronavirus contagious even incubation stage, China's health authority says. Published 26.1.2020, updated 27.1.2020, abgerufen am 27.1.2020
- ↑ Hoehl S. et al. Evidence of SARS-CoV-2 Infection in Returning Travelers from Wuhan, China, NEJM 2020
- ↑ Camilla Rothe, Mirjam Schunk et al. Transmission of 2019-nCoV Infection from an Asymptomatic Contact in Germany. NEJM 2020
- ↑ Menni et al. Symptom prevalence, duration, and risk of hospital admission in individuals infected with SARS-CoV-2 during periods of omicron and delta variant dominance: a prospective observational study from the ZOE COVID Study The Lancet 2022
- ↑ Dominic Wichmann et al. Autopsy Findings and Venous Thromboembolism in Patients With COVID-19. A Prospective Cohort Study Annals of Internal Medicine 2020
- ↑ ECDC Technical Report: Novel coronavirus (SARS-CoV-2) Discharge criteria for confirmed COVID-19 cases – When is it safe to discharge COVID-19 cases from the hospital or end home isolation?, 10.3.2020, abgerufen am 12.3.2020
- ↑ Lavezzo E. et al. Suppression of COVID-19 outbreak in the municipality of Vo Nature 2020