Translation (Biologie)
von lateinisch: translatio - Übertragung, Verlegung
Englisch: translation
Definition
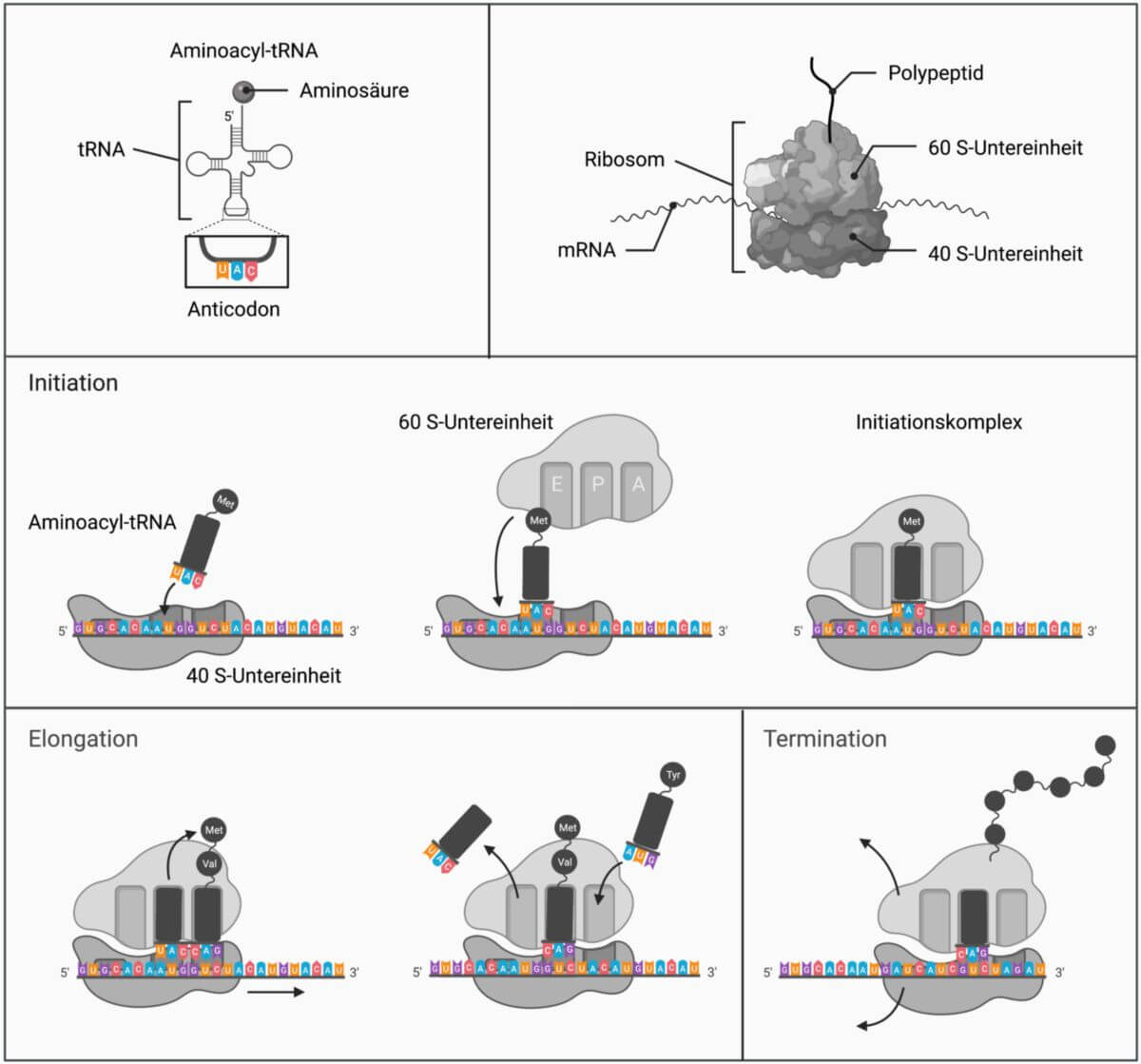
Die Translation ist ein Teilprozess der Proteinbiosynthese. Sie beinhaltet die Übersetzung der Basensequenz der Messenger-RNA (mRNA) in die Aminosäuresequenz der Proteine. An diesem Vorgang sind unter anderem Ribosomen und Transfer-RNAs (tRNAs) beteiligt.
Hintergrund
Die Übersetzung der Basensequenz in die Aminosäuresequenz basiert auf dem genetischen Code. Die Basenabfolge wird dabei in Dreiergruppen aufgeteilt, sogenannte Tripletts oder Codons. Da die mRNA aus vier verschiedenen Nukleotiden aufgebaut ist (Uracil (U), Cytosin (C), Adenin (A) und Guanin (G)) ergeben sich 43 = 64 mögliche Codons. Dies übersteigt die Anzahl der zu kodierenden 20 Aminosäuren, entsprechend werden einige Aminosäuren durch mehrere Codons kodiert. Es existieren drei Codons, die zum Abbruch der Translation führen, die Stopcodons UAG, UGA und UAA.
Als Adapter für die Translation von der mRNA zum Protein dient die tRNA. Sie erfüllen drei Funktionen:
- Träger der spezifischen Aminosäure
- Bindung an spezifische Codons der mRNA über das Anticodon
- Wechselwirkung mit Ribosomen
Trotz der hohen Spezifität existiert nicht für jedes Aminosäure-kodierende Codon eine individuelle tRNA. Die Wobble-Hypothese beschreibt das Phänomen, dass die Spezifität am 3'-Ende des Codons (und am 5'-Ende des Anticodons) geringer ist. Dadurch können beispielsweise die Alanincodons GCA, GCC und GCU alle von derselben tRNA gebunden werden.
Ablauf
Der Ablauf der Translation gliedert sich in drei Hauptphasen:
Zuvor müssen jedoch einige vorbereitende Reaktionen erfolgen.
Vorbereitung
Beladung der tRNAs
Die Beladung der tRNAs mit der korrekten Aminosäure wird durch Aminoacyl-tRNA-Synthetasen katalysiert. Diese Enzyme enthalten ein dreiteiliges aktives Zentrum, das die tRNA, die entsprechende Aminosäure und ein Adenosintriphosphat (ATP)-Molekül bindet. Unter ATP-Hydrolyse erfolgt in zwei Schritten die kovalente Verknüpfung der Aminosäure mit dem 3'-Ende der tRNA. Die Bindung ist energiereich und liefert zum späteren Zeitpunkt die Energie für die Synthese der Peptidbindung. Die beladene tRNA wird als Aminoacyl-tRNA bezeichnet.
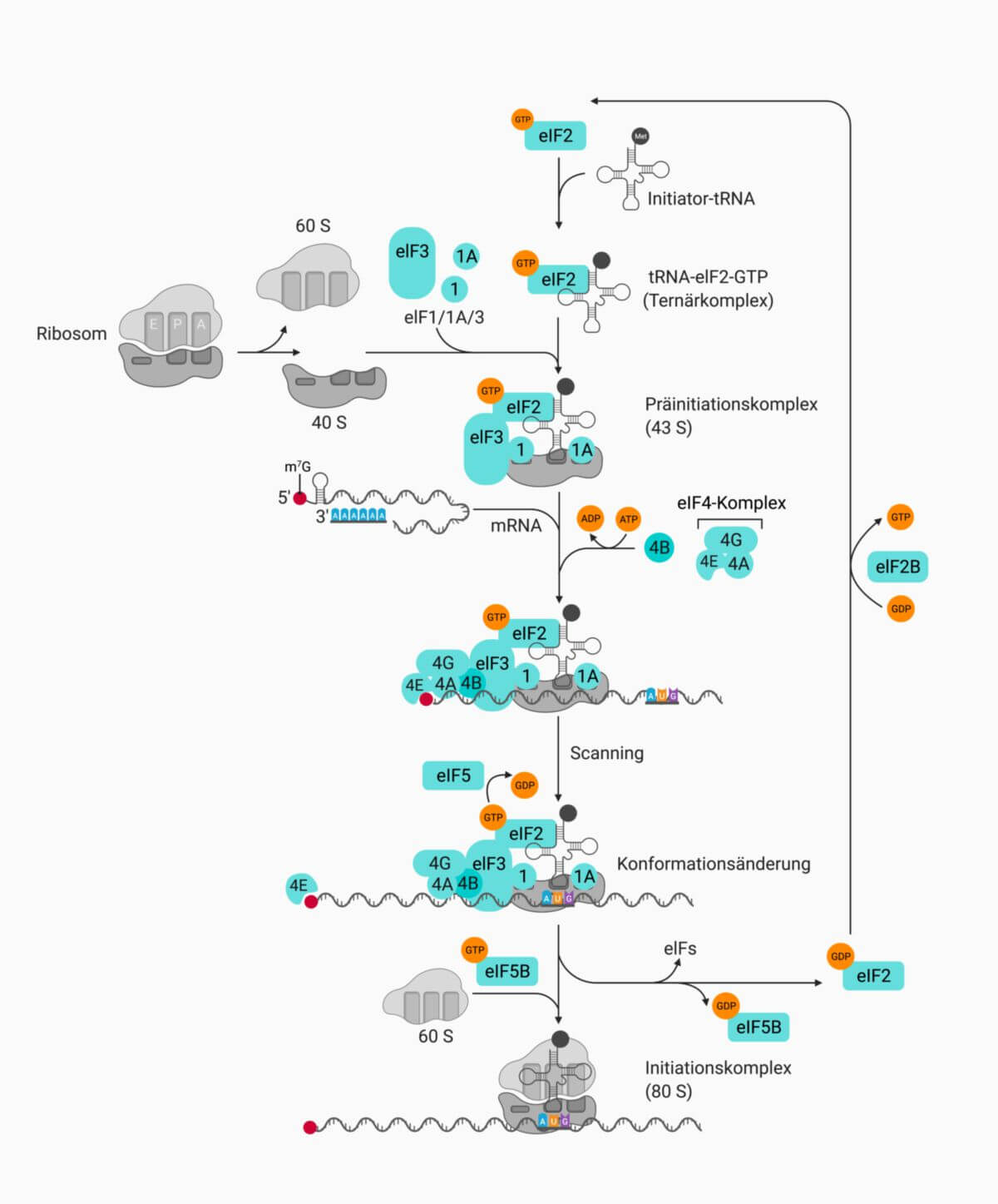
mRNA Aktivierung
Die Aktivierung der mRNA erfolgt ebenfalls unter ATP-Hydrolyse, wodurch die Bindung des eukaryotischen Initiationsfaktor (eIF) eIF4B und des eIF4F-Komplexes katalysiert wird.
Initiation
Die Translation beginnt mit der Bildung des Initiationskomplexes. Hierzu bindet zunächst die kleine ribosomale Untereinheit (40S-Untereinheit) an die aktivierte mRNA. Diese Interaktion erfordert die Präsenz von eIF3, eIF1 and eIF1A. Darauf folgt die Bindung der mit Methionin beladenen Initiator-tRNA im Komplex mit eIF2 und GTP.
Dieser Präinitiationskomplex (43 S) scannt die 5' untranslatierte Region (UTR) bis zum Startcodon (AUG), das sich meist im Kontext einer Kozak-Sequenz befindet. Hier kommt es zur Konformationsänderung der ribosomalen Untereinheit und zur GTP-Hydrolyse durch eIF5. Nachfolgend vermittelt eIF5B die Dissoziation von eIF1, eIF2-GDP und eIF1A sowie die konsequente Vereinigung mit der großen ribosomalen Untereinheit (60S-Untereinheit). Es entsteht der funktionale Initiationskomplex (80 S).
Elongation
Während der Elongation wandert das Ribosom am mRNA-Strang entlang und es binden sukzessive beladene tRNAs, deren Aminosäuren über Peptidbindungen verknüpft werden. Auf der großen Untereinheit des Ribosoms befinden sich drei Bindungsstellen, welche von den tRNAs nacheinander passiert werden:
- Aminosäure-Stelle (A-Stelle): hier findet die komplementäre Basenpaarung zwischen Codon und Anticodon statt
- Polypeptid-Stelle (P-Stelle): hier findet die Kopplung der Aminosäure an die wachsende Polypeptidkette statt
- Exit-Stelle (E-Stelle): hier verweilt die tRNA nach der Entladung der Aminosäure kurz, bevor sie zurück ins Zytosol diffundiert und erneut beladen werden kann
Zum Ende der Initiation befindet sich die Initiator-tRNA an der P-Stelle des Ribosoms. Eine beladene tRNA wird nun durch den eukaryotischen Elongationsfaktor (eEF) eEF1A zur A-Stelle befördert. Daraufhin fungiert die große Untereinheit als Peptidyltransferase, spaltet Methionin von der Initiator-tRNA ab und knüpft sie kovalent an die nachfolgende Aminosäure. Die Translokation des Ribosoms findet unter eEF2-vermittelter GTP-Hydrolyse statt.
Termination
Sobald ein Stopcodon an die A-Stelle tritt, wird die Elongation terminiert. Es existiert keine passende tRNA, stattdessen binden sogenannte eukaryotische Releasefaktoren (eRF), eRF1 und eRF3, welche die Bindung zwischen dem Polypeptid und der tRNA an der P-Stelle hydrolysieren.
Posttranslationale Ereignisse
Schon während der Translation findet die Faltung des Proteins statt. Die Sekundär- und Tertiärstruktur werden dabei durch die Aminosäuresequenz bestimmt und Chaperone unterstützen die korrekte Faltung. Weiterhin können posttranslationale Modifikationen (z.B. Glykosylierung oder Phosphorylierung) sowie Prozessierungen (z.B. Proteolyse) notwendig sein, damit das Protein seine Funktionalität erlangt.
Unterschiede bei Prokaryoten
Grundsätzlich ist der Ablauf der Translation bei Prokaryoten dem der Eukaryoten sehr ähnlich und gliedert sich ebenfalls in Initiation, Elongation und Termination. Wesentliche Unterschiede bestehen in den beteiligten Molekülen, sowie der Erkennung der mRNA. Das prokaryotische Ribosom (70 S) wird durch die kleine Untereinheit (30 S) und die große Untereinheit (50 S) gebildet. Die kleine Untereinheit bindet die mRNA über die sogenannte Shine-Dalgarno-Sequenz, die sich upstream zum Startcodon befindet. An der Bildung des Initiationskomplexes sind die drei prokaryotischen Initiationsfaktoren IF-1, IF-2 und IF-3 beiteiligt. Die Elongations- und Releasefaktoren haben andere Namen, arbeiten aber prinzipiell nach demselben Schema wie die eukaryotischen.
Lokalisation
Die Translation kann entweder im Zytosol stattfinden oder am endoplasmatischen Retikulum (ER). Befindet sich zu Beginn des Polypeptides ein Signalpeptid mit einer spezifischen Aminosäuresequenz, vermittelt dieses die Bindung an einen Signalerkennungspartikel (SRP). Dieser Komplex blockiert zunächst die weitere Elongation und rekrutiert das Ribosom an das raue ER. Die Verteilung zum Bestimmungsort erfolgt dann über das Endomembransystem.
Polysome
Bei der Translation können mehrere Ribosomen gleichzeitig aktiv sein. So bildet sich ein neuer Initiationskomplex, nachdem das erste Ribosom bereits einen Teil an der mRNA entlang gewandert ist. Unter dem Elektronenmikroskop erscheinen diese Polysomen als perlenkettenartige Aufreihung an der mRNA.
Klinik
Einige Bakterien produzieren Toxine, welche die Translation in menschlichen Zellen beeinträchtigen. Beispiele sind das Diphtherietoxin von Corynebacterium diphtheriae und Exotoxin A von Pseudomonas aeruginosa.
Pharmakologie
Die Translation bestimmter Proteine kann durch Antisense-Oligonukleotide gezielt unterbunden werden. Beispiele sind Inotersen oder Volanesorsen.
Diverse Antibiotika hemmen die bakterielle Translation und verringern oder unterbinden damit die Vermehrung. Hierzu zählen u.a.:
Quellen
- Jackson et al. The mechanism of eukaryotic translation initiation and principles of its regulation Nat Rev Mol Cell Biol 2010
- Guerrero et al. HIV-1 Replication and the Cellular Eukaryotic Translation Apparatus Viruses 2015
Literatur
- Purves, Sadava et al., Biologie, 7. Auflage, 2006, Spektrum