Proteasom
Synonym: 26S-Proteasom
Englisch: proteasome
Definition
Proteasomen sind zelluläre Proteinkomplexe, die als multikatalytische Protease fungieren. In eukaryotischen Zellen sind sie als Bestandteil des Ubiquitin-Proteasom-Systems für den Teil des Proteinabbaus verantwortlich, der außerhalb der Lysosomen stattfindet.
Geschichte
Für die Entdeckung des Ubiquitin-gesteuerten Proteinabbaus erhielten Aaron Ciechanover, Avram Hershko und Irwin Rose 2004 den Nobelpreis für Chemie.
Vorkommen
Proteasomen kommen bei allen Eukaryoten und Archaeen sowie bei einigen Bakterien vor. In eukaryotischen Zellen sind sie im Zytoplasma und im Zellkern zu finden.
Biochemie
Proteasomen sind zusammen mit den Lysosomen die Orte für die intrazelluläre Qualitätskontrolle und für den Abbau von nicht mehr benötigten Proteinen.
Proteasomen bauen diese Proteine unter ATP-Verbrauch ab. Dazu müssen die Substrate vorher durch das Anhängen von Polyubiquitinketten markiert werden (Ubiquitinsystem). Anschließend gelangen die Proteine zum Proteasom, werden entfaltet, zu Peptiden aus 7-9 Aminosäuren gespalten und wieder aus dem Proteasom freigesetzt.
Struktur
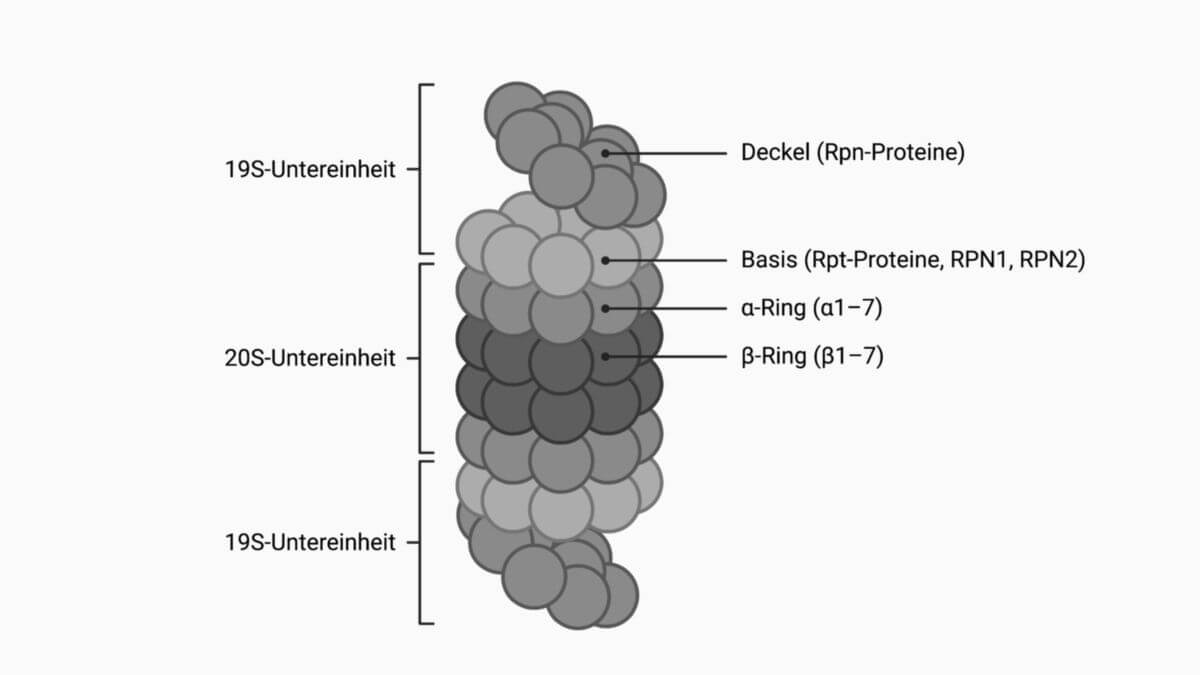
Eukaryotische Proteasomen werden basierend auf der Sedimentationskonstante als 26S-Proteasom bezeichnet. Sie besitzen ein Molekulargewicht von ca. 2.000 kDa sowie eine Größe von 18 x 11 nm und bestehen aus drei Komponenten:
- einer katalytischen 20S-Untereinheit und
- zwei regulatorischen 19S-Untereinheiten
Dem Proteasom von Prokaryoten fehlen die 19S-Untereinheiten.
20S-Untereinheit
Die 20S-Untereinheit bildet den Hauptteil des Proteasoms und besteht aus vier gestapelten Proteinringen mit wiederum jeweils sieben Untereinheiten. Das 20S-Partikel hat dadurch die Form eines Zylinders und weist insgesamt ein Molekulargewicht von ca. 750 kDa auf.
Die Untereinheiten der beiden äußeren Ringe werden als α1 bis α7, die der inneren Ringe als β1 bis β7 bezeichnet. Die beiden inneren Ringe stellen das aktive Zentrum des Enzyms dar. Dabei besitzen die Untereinheiten β1, β2 und β5 eine katalytische Aktivität und schneiden spezifisch die Peptidbindung zwischen verschiedenen Aminosäuren:
- β1: Spaltung der Peptidkette nach sauren Aminosäuren (Caspase-ähnliche Aktivität, auch Peptidylglutamyl-Peptid hydrolysierende bzw. PGPH-Aktivität bezeichnet)
- β2: Spaltung der Peptidkette nach basischen Aminosäuren (Trypsin-ähnliche Aktivität)
- β5: Spaltung der Peptidkette nach hydrophoben Aminosäuren (Chymotrypsin-ähnliche Aktivität)
Die beiden äußeren Ringe haben regulierende Aufgaben. Sie verhindern den unkontrollierten Zugang von Substraten zum inneren Hohlraum des Proteasoms.
19S-Untereinheit
Die 19S-Untereinheiten fungieren als Verschluss der 20S-Untereinheit und lagern sich an die beiden Öffnungen an. Sie bestehen aus je 19 Proteinen, die zusammen mindestens 20 Konformationszustände einer 19S-Untereinheit bewirken können. Man unterscheidet grundsätzlich zwischen einer Basis und einem Deckel.[1]
Die Basis beinhaltet sog. Rpt-Proteine (Rpt1 bis Rpt6), die sich jeweils zu einem Heterodimer zusammenlegen und insgesamt einen hexameren Ring ausbilden. Sie fungieren als AAA-ATPasen. Die ATP-Hydrolyse ist notwendig für die Entfaltung der Zielproteine.
Alle weiteren Proteine werden als Rpn-Proteine bezeichnet, z.B.:
- Rpn13: Ubiquitinrezeptor
- Rpn10 und 11: deubiquinierende Enzyme, die Ubiquitin-Einheiten direkt vor der Translokation von Substraten in die 20S-Untereinheit entfernen.
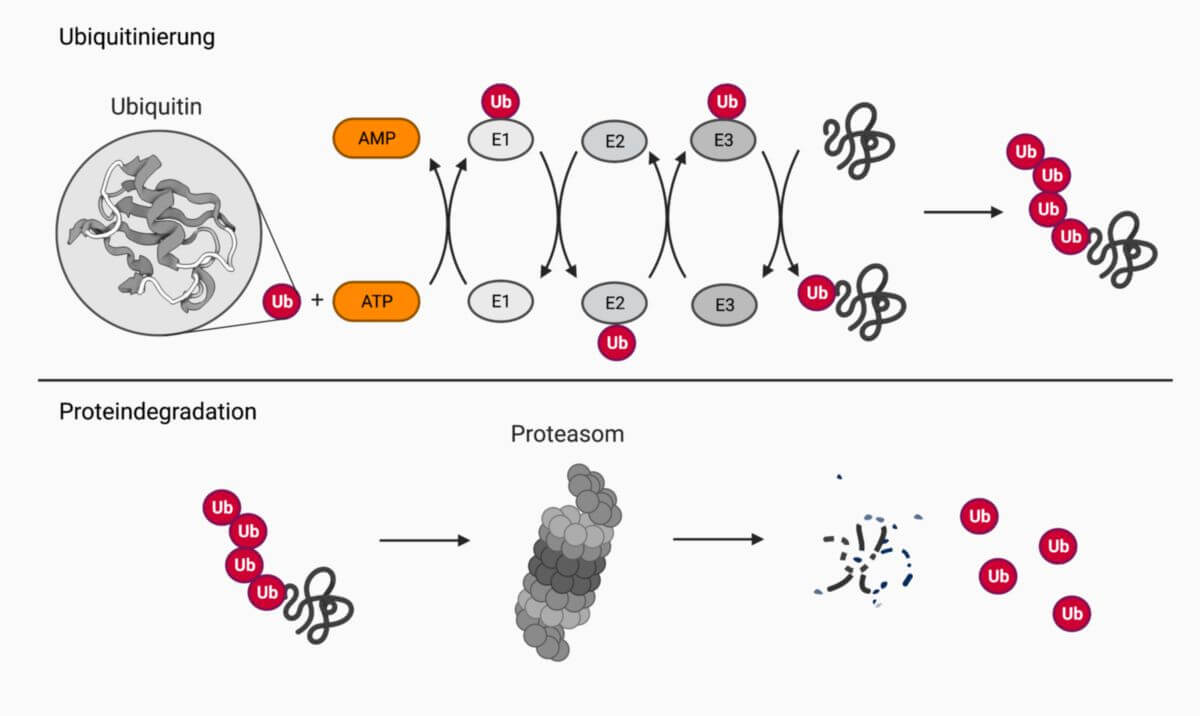
Ubiquitinsystem
Ubiquitin ist ein kleines, hoch konserviertes Protein aus 76 Aminosäuren. Die Markierung der abzubauenden Proteine wird durch ein komplexes Enzymsystem durchgeführt. Man unterscheidet:
- Ubiquitin-aktivierende Enzyme (E1): ATP-Hydrolyse und Bindung von Ubiquitin über eine Thioesterbindung
- Ubiquitin-konjugierende Enzyme (E2): Ubiquitin wird von E1 auf eine SH-Gruppe von E2 übertragen. Das E2-Enzym bildet einen Komplex mit E3 und überträgt Ubiquitin auf letzteres Enzym.
- Substraterkennungsproteine (E3): Erkennen das Substrat und transferieren Ubiquitin von E2 auf das Zielprotein.
Diese Reaktionen laufen mehrmals hintereinander ab, wobei das nächste Ubiquitinmolekül auf das vorhergehende übertragen wird, sodass eine Polyubiquitinkette entsteht. Ein Zielprotein muss mit mindestens vier Ubiquitinmonomeren markiert sein, bevor es in einem ATP-abhängigen Schritt vom Deckel der 19S-Untereinheit erkannt wird.
Einige Proteine werden Ubiquitin-unabhängig im Proteasom abgebaut, z.B. Ornithin-Decarboxylase.
Translokation
Nachdem das Zielprotein von der 19S-Untereinheit erkannt wurde, muss es in das Innere des 20S-Partikels gelangen. Da der zentrale Kanal eng ist, muss das Substrat zumindest um ca. 20 Aminosäurereste ATP-abhängig entfaltet werden. Der Durchgang des ungefalteten Substrats in den Kern wird als Translokation bezeichnet. Sie erfolgt nach einer Deubiquitinierung und ohne ATP-Spaltung. Die Translokation ist jedoch erst möglich, wenn die 19S-Kappe ATP gebunden hat.
Proteolyse
Das Proteasom fungiert als Endoprotease, wobei die Proteolyse durch die β-Untereinheiten des 20S-Partikels auf einem Threonin-abhängigen nukleophilen Angriff beruht. Der Proteinabbau in der zentralen Kammer führt zur Freisetzung von kurzen Oligopeptiden aus 7-9 Aminosäuren.
Zum Teil sind die kurzen Peptidfragmente biologisch aktiv. So werden einige Transkriptionsfaktoren als inaktive Vorstufe synthetisiert und erst durch proteasomalen Abbau aktiviert.
Funktion
Das Proteasom baut Proteine ab, die in der Zelle nicht mehr gebraucht werden. Dazu gehören u.a. folgende Proteine:
- regulatorische Proteine, die inaktiviert werden müssen: z.B. Cycline im Zellzyklus
- falsch gefaltete Proteine (z.B. Prionen), die von Chaperonen gebunden werden und sonst akkumulieren würden.
- nicht funktionsfähige Proteine (z.B. aufgrund von Mutationen)
- virale Proteine, die anschließend über MHC-Klasse-I-Komplexe Immunzellen präsentiert werden (Immunproteasom)
Das Proteasom spielt zusammen mit den Caspasen eine wichtige Rolle bei der Apoptose.
Immunproteasom
Im Rahmen einer Immunantwort exprimieren einige hämatopoetische Zellen als Reaktion auf proinflammatorische Signale (z.B. Zytokine) alternative katalytische β-Untereinheiten:
- β1i: LMP2 (low molecular mass protein 2)
- β2i: MECL-1 (multicatalytic endopeptidase complex like 1)
- β5i: LMP7 (low molecular mass protein 7)
Interferon-γ sorgt außerdem dafür, dass sich die katalytische 20S-Untereinheit mit einer regulatorischen 11S- statt 19-Untereinheit verbindet.
Das so zusammengesetzte Proteasom wird als Immunproteasom bezeichnet. Es besitzt besondere Funktionen für die Immunantwort, beispielsweise ermöglicht es eine stärkere Präsentation von Antigenen über MHC-Klasse I im Vergleich zum normalen ("konstitutiven") Proteasom. In einigen Immunzellen (z.B. Lymphozyten) wird das Immunproteasom dauerhaft gebildet.
Thymomproteasom
In kortikalen Epithelzellen des Thymus kommt eine weitere Sonderform des Proteasoms vor, das Thymomproteasom. Dabei ist die Untereinheit β5 durch β5t ersetzt. Es ist beteiligt an der positiven Selektion von T-Lymphozyten.
Α4-α4-Proteasom
Das α4-α4-Proteasom ist eine alternative Isoform, die insbesondere bei Hefen, aber auch in einigen menschlichen Zellen vorkommt.[2] Bei dieser Variante ist die α3- durch eine α4-Untereinheit ausgetauscht. Die genaue Funktion ist derzeit (2025) unbekannt, es scheint jedoch mit einer erhöhten Resistenz gegen toxische Metallionen (z.B. Cadmium) einherzugehen.
Klinik
Störungen des Proteasoms und des Ubiquitinsystems spielen eine pathogenetische Rolle bei verschiedenen Erkrankungen, z.B. bei:
Pharmakologie
Die Hemmung des Proteasoms hat unterschiedliche Auswirkungen auf die Apoptose in verschiedenen Zelllinien, in schnell teilenden Zellen wirkt sie meist proapoptotisch. Dieser Effekt wird pharmakologisch mittels Proteasom-Inhibitoren (z.B. Bortezomib) ausgelöst.
Quellen
- ↑ Beck F et al. Near-atomic resolution structural model of the yeast 26S proteasome, PNAS September 11, 2012 109 (37) 14870-14875, abgerufen am 08.04.2020
- ↑ Padmanabhan A et al. Assembly of an Evolutionarily Conserved Alternative Proteasome Isoform in Human Cells, Cell Reports. 14 (12): 2962–74. March 29, 2016, abgerufen am 08.04.2020