Spleißen
von englisch: to splice - verbinden, zusammenfügen
Synonym: Splicing
Englisch: splicing
Definition
Als Spleißen wird ein Vorgang bei der RNA-Prozessierung bezeichnet, bei dem die Introns aus der transkribierten hnRNA-Kette entfernt werden. Er wird durch einen RNA-Protein-Komplex, das sogenannte Spleißosom, katalysiert. Das Spleißen ist entscheidend für die Bildung der reifen mRNA.
Hintergrund
Eukaryotische Gene bestehen aus kodierenden und nicht kodierenden Abschnitten, sogenannten Exons und Introns. Bei der Transkription wird zunächst die hnRNA gebildet, die sowohl Exons als auch Introns enthält. Im Zellkern finden daraufhin verschiedene RNA-Prozessierungsschritte statt. Hierzu zählen neben dem Spleißen das Capping und die Polyadenylierung.
Ablauf
Basismechanismus
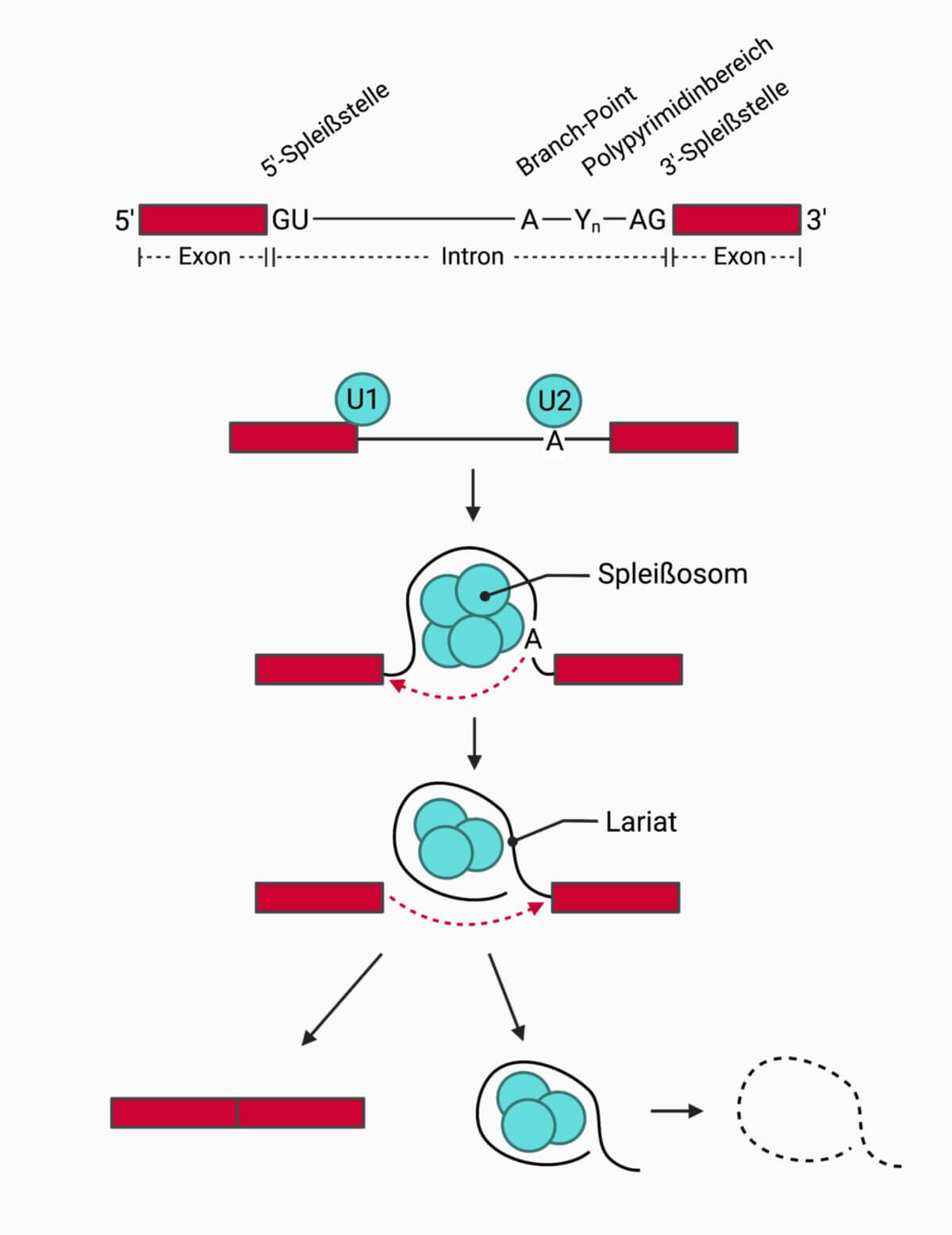
An den Grenzen zwischen Introns und Exons befinden sich Konsensussequenzen, die als Erkennungssignal für verschiedene snRNPs ("small nuclear ribonucleoproteins") dienen. Das U1 snRNP bindet mit seinem RNA-Anteil über komplementäre Basenpaarung an die 5'-Spleißstelle. Das U2 snRNP lagert sich an den Branch-Point an, einen Bereich nahe der 3'-Exon-Intron-Grenze, der durch ein konserviertes Adenin gekennzeichnet ist.
Unter Energieverbrauch aus ATP-Hydrolyse lagern sich weitere snRNPs an, die gemeinsam das Spleißosom bilden. Es kommt zur Spaltung an der 5'-Spleißstelle, woraufhin das Intron eine Lassostruktur (Lariat) bildet. Das resultierende freie 3'-Ende des Exons greift daraufhin an der 3'-Exon-Intron-Grenze an und wird mit dem nachfolgenden Exon verknüpft.
Bei beiden Reaktionen handelt es sich um eine nukleophile Substitution (SN2-Reaktion). Dabei greift jeweils eine nukleophile Hydroxygruppe an einem Phosphat an.
Alternatives Spleißen
Aus hnRNAs können durch Variationen beim Spleißen verschiedene reife mRNA-Varianten hervorgehen. Somit können aus einem Gen mehrere funktionelle Proteine gebildet werden. Dies ist eine evolutiv bedeutsame Entwicklung bei Eukaryoten und erleichtert die Anpassung an veränderte Lebensbedingungen.
siehe Hauptartikel: Alternatives Spleißen
Autospleißen (Selbstspleißen)
In einigen Organismen existieren katalytisch wirksame RNAs, sogenannte Ribozyme, die in der Lage zum Autospleißen sind. Sie besitzen eine charakteristische Sekundär- und Tertiärstruktur, die ihnen ermöglicht, das Spleißen ohne die Hilfe zusätzlicher Proteine durchzuführen. Dieser Vorgang wurde zuerst bei der 26S-rRNA von Ciliaten beschrieben.
Abhängig vom Mechanismus des Autospleißens wird zwischen Gruppe-I- und Gruppe-II-Introns unterschieden. Der Spleißvorgang bei Gruppe-II-Introns ähnelt dem Spleißosom-vermittelten Spleißen und wird daher als eventueller phylogenetischer Vorläufer vermutet.
Klinik
Genmutationen in Introns können das korrekte Spleißen beeinträchtigen und somit z.B. zur Intronretention oder zum Exonskipping führen. Solche Spleißmutationen sind u.a. als Auslöser für Mukoviszidose und die Muskeldystrophie Typ Duchenne beschrieben.