Spliceosom
Synonym: Spleißosom
Englisch: spliceosome
Definition
Das Spliceosom ist ein großer, im Zellkern gelegener, RNA-Protein-Komplex, der den Spleißvorgang bei der RNA-Prozessierung katalysiert. Er besteht aus fünf verschiedenen kleinen RNA-Molekülen (snRNAs) und mehr als 100 verschiedenen Proteinen.[1]
Vorkommen
Das Spliceosom konnte sowohl bei niederen Organismen wie Hefe, als auch bei Säugetieren nachgewiesen werden.
Struktur
Das Spliceosom hat eine Molekülmasse von mehreren Megadaltons. Die Größe ist beim Menschen vergleichbar mit der 60 S-Untereinheit der Ribosomen.[2]
Der Komplex besteht aus den snRNPs ("small nuclear ribonucleoproteins") U1, U2, U4, U5 und U6. Die snRNPs sind wiederum aus snRNAs und den sieben Sm-Proteinen B/B’, D1, D2, D3, E, F und G zusammengesetzt. Dies sind die Kernproteine der snRNPs. Die Struktur des Spliceosoms ist sehr dynamisch. Während dem Spleißen bilden sich aus den einzelnen snRNPs mehrere Subkomplexe, beispielsweise der U4-U5-U6-Komplex. Diese enthalten neben den Sm-Proteinen noch zahlreiche weitere Proteinkomponenten, unter anderem die LSm-Proteine (von "like-Sm").
Funktion
Mechanismus
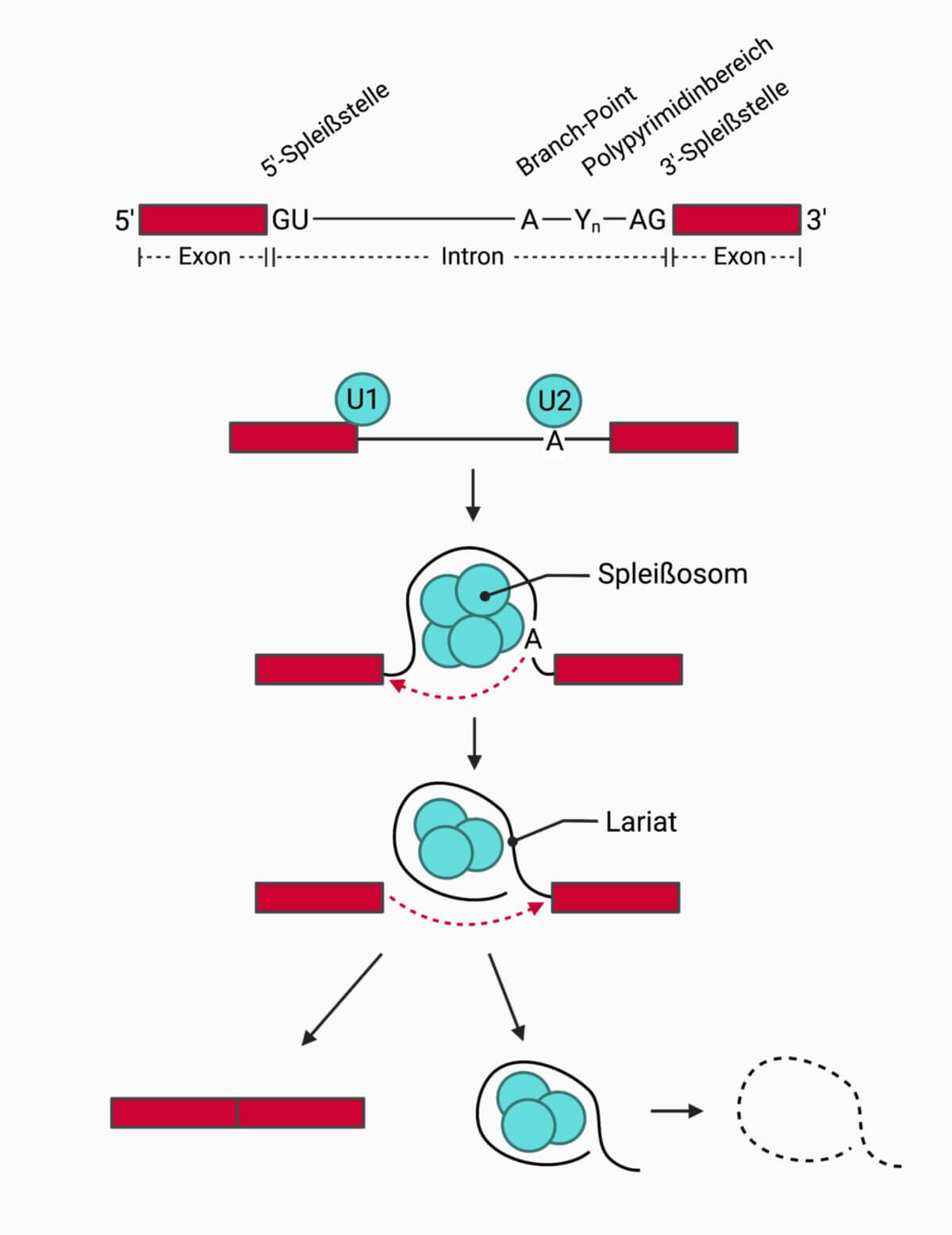
Intronstruktur
Ein Intron ist durch folgende strukturellen Merkmale gekennzeichnet:
- Das Introns beginnt mit dem Dinukleotid GU (Donorsequenz).
- Das Introns endet mit dem Dinukleotid AG (Akzeptorsequenz).
- Der sogenannte Branch-Point mit einem konservierten Adenin befindet sich ca. 18–40 Nukleotide upstream der 3'-Spleißstelle.
- Bei Säugetieren schließt sich an den Branch-Point noch ein Polypyrimidinbereich an.
Das eigentlichen Spleißen besteht aus zwei aufeinanderfolgenden Transesterifikationen (Umesterungen), welche durch die snRNPs katalysiert wird.
Bildung des Spliceosom
Das Spliceosom bildet sich, wenn das U1 snRNP an die 5'-Spleißstelle GU bindet und U2 an den Branch-Point. Diese Bindung ist möglich, da die snRNA im snRNP-Molekül komplementäre Sequenzen besitzt. Der U2-BP-Komplex wird auch als "A-Komplex" oder "Prä-Spliceosom" bezeichnet.
Die snRNPs U4, U5, U6 bilden schon vor ihrer Bindung einen Tri-snRNP-Komplex aus. Sobald dieser zur 5'-Spleißstelle rekrutiert wird, trägt er den Namen "B-Komplex". Diese Interaktion führt zu einer Strukturveränderung der prä-mRNA, wodurch die gebundenen snRNPs U1 und U4 destabilisiert werden und sich ablösen. U6 befindet sich nun an der ehemaligen Position von U1. Durch diese Reaktion befindet sich das Spliceosom in einem katalytisch aktiven Zustand.
Erste katalytische Reaktion
Durch die Verformung der prä-mRNA befinden sich nun U6 und U2 in räumlicher Nähe und sind durch Interaktion ihrer snRNAs aneinander gebunden. Dies ermöglicht den nukleophilen Angriff des Adenins aus dem Branch-Point (A-Komplex) auf die 5'-Spleißstelle. Die gebildete Loop-Struktur wird als Lariat bezeichnet.
Zweite katalytische Reaktion
Die U5-Untereinheit bringt die beiden Exons in räumliche Nähe zueinander. Dadurch kann der zweite nukleophile Angriff durch die freie 3'-Hydroxylgruppe erfolgen, der die Exons miteinander verbindet. Das Lariat wird dadurch abgetrennt und nachfolgend abgebaut. Die snRNPs werden recycled und stehen erneut zur Verfügung.[3]
Quellen
- ↑ Basiswissen Humangenetik" - Christian P. Schaaf, Johannes Zschocke, Springer-Verlag, 2. Auflage
- ↑ Klug, W. S. Concepts of genetics. 10th edn, (Pearson Education, 2012).
- ↑ Will, C. L. & Luhrmann, R. Spliceosome structure and function. Cold Spring Harb Perspect Biol 2011