DNA-Reparatur
Englisch: DNA repair
Definition
Unter DNA-Reparatur versteht man verschiedene biochemische Mechanismen, die mithilfe von Enzymen durch endogene oder exogene Faktoren entstandene DNA-Schäden beseitigen. Diese Reparatur ist für eine fehlerfreie DNA-Replikation unabdingbar, da es während der Replikation häufig zu Fehlern durch die beteiligten Enzyme kommt.
siehe auch: DNA-Schaden, DNA Damage Response
Reparaturmechanismen
Proofreading
Ein Reparaturmechanismus, der bei der DNA-Replikation zum Tragen kommen kann, ist das sogenannte Proofreading (engl. für Korrekturlesen). Beim Menschen korrigieren nur die Polymerasen δ und ε (und γ in den Mitochondrien) in 3'-5'-Richtung direkt Fehler, die sie selber verursacht haben. Die Replikationsgeschwindigkeit liegt beim Menschen daher bei 50 Nukleotiden pro Sekunde, verglichen mit ca. 1.000 Nukleotiden pro Sekunde bei Bakterien, bei denen alle Polymerasen zum Proofreading fähig sind.
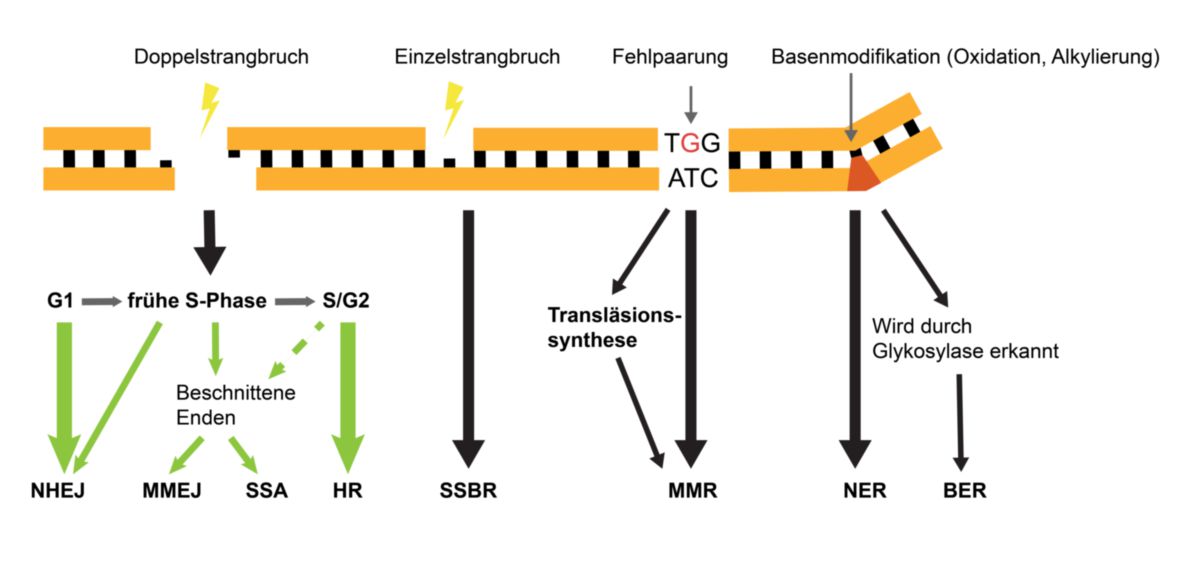
Doppelstrangbruch
Ein DNA-Doppelstrangbruch ist die kritischste Beschädigung der DNA, da die Gefahr weitreichender Chromosomenumlagerungen besteht und die Reparatur oft fehlerbehaftet ist. Es existieren mehrere Reparaturmechanismen, die der Zelle zur Verfügung stehen und vom Zellzyklus und der Art, wie die freien DNA-Enden beschnitten werden, abhängig sind.
Nichthomologe-Endverknüpfung
Die Nicht-homologe Endverknüpfung (NHEJ) ist der häufigste Reparaturmechanismus für Doppelstrangbrüche und sequenzunabhängig. Er fügt DNA-Enden zusammen, unabhängig davon ob Sequenzen verloren gegangen sind, und gilt daher als fehlerbehaftet. Die NHEJ steht der Zelle in jeder Phase des Zellzyklus zur Verfügung, ist jedoch inhibiert, sobald die Bruchstellen beschnitten werden. Daher ist die NHEJ in der G1-Phase am aktivsten.
Homologe Rekombination
Die homologe Rekombination ist der einzige fehlerfreie Reparaturmechanismus für Doppelstrangbrüche. Er verwendet ein Schwesterchromatid als Vorlage um homologe 3'-Überhänge zu erzeugen, die sich danach wieder anlagern können. Aufgrund der Abhängigkeit vom Vorliegen der Schwesterchromatiden ist die homologe Rekombination auf die späte S-Phase und G2-Phase beschränkt.
MMEJ
Microhomology-mediated end joining (MMEJ) verwendet kurze homologe Sequenzen (5-25 bp) auf beiden Seiten der Bruchstelle, um die Stränge wieder zu verbinden. Die DNA wird soweit zurückgeschnitten, bis diese Homologien freigelegt sind. Da hierbei sehr große Deletionen entstehen können, besitzt der Mechanismus ein sehr großes Mutationspotential. Gelegentlich wird er daher auch als Back-Up-Prozess charakterisiert, wenn die Reparatur mit anderen Mechanismen nicht möglich ist.
Single-Strand Annealing
Single-Strand Annealing (SSA) zeigt viele Ähnlichkeiten zu MMEJ. Die Stränge werden ebenfalls bis zu einer homologen Sequenz zurückgeschnitten, jedoch bestehen diese Sequenzen aus Tandem Repeats. Wie beim MMEJ können dadurch sehr große Deletionen entstehen. Es wird diskutiert, ob es sich beim SSA nicht um eine Unterform des MMEJ handelt. Obwohl einige Proteine in beiden Mechanismen vorkommen, tendieren die meisten Studien mittlerweile dazu, das SSA als eigenständigen Prozess zu betrachten.
Einzelstrangbruch
Ein Einzelstrangbruch kann als natürliches Zwischenprodukt zellulärer Vorgänge oder durch Mutagene entstehen. Ein Einzelstrangbruch tritt sehr häufig in der Zelle auf und kann fast immer zuverlässig repariert werden. Freie DNA-Enden werden durch die Zelle erkannt. Der wichtigste Schritt der Reparatur ist die Erzeugung von kompatiblen 5‘- und 3‘-Enden, die durch eine DNA-Polymerase und eine DNA-Ligase wieder verbunden werden können.
Fehlpaarungsreparatur
Die Fehlpaarungsreparatur erkennt Fehlpaarungen und entfernt diese durch Exonukleasen. Die Lücke wird durch DNA-Polymerasen und DNA-Ligasen verschlossen. Die Besonderheit an diesem Mechanismus ist, dass die Zelle unterscheiden kann, welcher der neusynthetisierte fehlerhafte DNA-Strang ist.
Transläsionssynthese
Trifft eine DNA-Polymerase während der Replikation auf ein Hindernis in der DNA (Basenmodifikation, Pyrimidin-Dimer) kann dies zum Zusammenbruch der Replikationsgabel führen. Dies kann durch die Transläsionssynthese verhindert werden. Für wenige Nukleotide wird die DNA-Polymerase durch eine weniger zuverlässige DNA-Polymerase getauscht. Diese kann trotz des Hindernisses die DNA-Synthese fortsetzen. Nach einigen Nukleotiden wird sie wieder durch die normale DNA-Polymerase ersetzt.
Fanconi-Anämie-Pathway
Der Fanconi-Anämie-Pathway repariert zytotoxische Interstrand Crosslinks (ICLs), die durch Crosslinker in die DNA eingeführt wurden. Er ist einer der komplexesten Reparaturmechanismen der Zelle, da er Charakteristiken der homologen Rekombination, Nukleotid-Exzisionsreparatur und Transläsionssynthese aufweist.
Basenmodifikationen
Eine chemisch modifizierte Nukleobase kann innerhalb der DNA zu Punktmutationen durch Transversionen führen. Verbleibt sie bis zur Replikation unrepariert, kann dies zum Zusammenbruch der Replikationsgabel und zu Doppelstrangbrüchen führen. Es existieren verschiedene Reparaturmechanismen.
Nukleotidexzisionsreparatur
Die Nukleotidexzisionsreparatur (NER) ist einer der wichtigsten Reparaturmechanismen der Zelle. Er erkennt keine spezifische Beschädigung, sondern Veränderungen in der DNA-Konformation, wie sie durch modifizierte Basen oder DNA-Addukte wie Pyrimidin-Dimere entstehen. Unabhängig von der Art des Schadens wird immer ein 24-32 Nukleotide langes Segment aus dem beschädigten Strang entfernt. Die Lücke wird durch DNA-Polymerasen und DNA-Ligasen verschlossen.
Basenexzisionsreparatur
Zahlreiche Basenmodifikationen werden durch die Basenexzisionsreparatur (BER) korrigiert. Glykosylasen erkennen spezifische Modifikationen und entfernen die beschädigte Base. Es entsteht kurzfristig eine abasische Stelle (Englisch: "abasic site"). Beschädigungen für die keine Glykosylase existieren, werden nicht repariert.
Direkte Entfernung der Basenmodifikation
Einige Modifikationen wie O-6-Methylguanin (Methylierung von Guanin) können auch direkt rückgängig gemacht werden. Das Enzym O-6-Methylguanin-DNA-Methyltransferase entfernt hier die zusätzliche Methylgruppe, ohne die Base zu entfernen.
Photoreaktivierung
Pyrimidin-Dimere können durch einen weiteren Mechanismus aufgelöst werden, die Photoreaktivierung. Eine Photolyase wird durch Licht angeregt und überträgt temporär ein Elektron auf das Dimer, wodurch die Bindung aufgebrochen wird. Dieser Mechanismus existiert aber nur in niederen Organismen, der Mensch besitzt keine Photolyasen.
Medizinische Relevanz
Gestörte DNA-Reparaturmechanismen führen zu fehlerhafter DNA-Replikation und zur Anfälligkeit gegenüber mutagenen Einflüssen, die mit dem natürlichen Alterungsprozess, aber auch mit der Entstehung von Tumoren assoziiert sind. Beipiel für eine assoziierte Krankheit ist Xeroderma pigmentosum, die auf ein fehlerhaftes Reparatursystem zurückzuführen ist. Hier kommt es vermehrt zu Schäden durch UV-Strahlung in der Haut und durch die fehlende Reparatur zu einer starken Erhöhung der Tumorneigung.