Pyrimidin-Dimer
Definition
Als Pyrimidin-Dimere werden kovalent verknüpfte Pyrimidinreste innerhalb der DNA bezeichnet. Sie entstehen durch UV-Strahlung und können zu weiteren Mutationen im Genom führen.
Entstehung
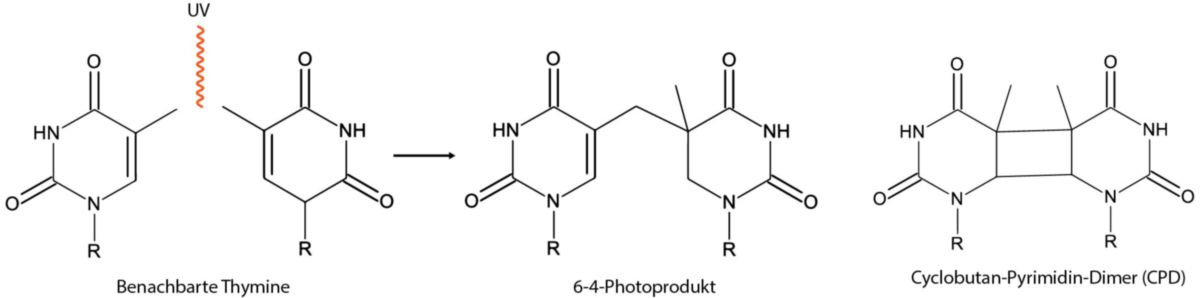
Die Hauptursache für Pyrimidin-Dimere ist UV-Strahlung. Pyrimidine absorbieren besonders UV-Strahlung aus dem UV-B-Bereich (280-320 nm). Die Energie der UV-Strahlung wird von der Doppelbindung im Pyrimidinring absorbiert und ermöglicht es, Reaktionen mit benachbarten Molekülen einzugehen. Die häufigste daraus entstehende Struktur wird als Cyclobutan-Pyrimidin-Dimer (CPD) bezeichnet. Es kann aber auch zur Ausbildung eines 6-4-Photoprodukts kommen. Dieses ist nur über eine einzelne kovalente Bindung miteinander verbunden. Thymin-Dimere sind deutlich häufiger zu beobachten als Cytosin- oder Cytosin/Thymin-Dimere.[1]
Auswirkungen
Pyrimidin-Dimere führen zu einer Störung in der DNA-Konformation. Dies stellt ein Hindernis während der Replikation dar und führt unrepariert zum Stopp oder sogar Zusammenbruch der Replikationsgabel.
Die Zelle besitzt aber auch die Möglichkeit diese Nukleotide einfach zu überspringen, wodurch eine Lücke im neu-synthetisierten DNA-Strang entsteht. Diese Lücke kann durch die Postreplikative Reparatur aufgefüllt werden. Das Dimer bleibt bei diesem Mechanismus aber bestehen und muss durch andere Reparaturmechanismen korrigiert werden.
Besonders Thymin-Dimere können auch durch Transläsionssynthese erkannt werden, wodurch auf dem neu-synthetisierten Strang entsprechend Adenin eingebaut wird. Jedoch kann es während dieser Replikation auch zu Fehlern kommen. Am häufigsten werden hier Cytosin-Dimere von der Polymerase als Thymine erkannt und am neu-synthetisierten Strang werden entsprechend Adenine eingebaut. Hier kommt es zu einer Cytosin-zu-Thymin-Transversion.[2][3]
Reparatur
Störungen in der DNA-Konformation werden durch die Nukleotid-Exzisionsreparatur (NER) repariert. Ein Proteinkomplex erkennt die Strukturveränderung und rekrutiert Nukleasen, die ein gesamtes Segment (ca. 28 Nukleotide in Eukaryoten) mit dem Schaden komplett herausschneiden. Diese Lücke wird durch DNA-Polymerasen und Ligasen wieder verschlossen.
Escherichia coli besitzt außerdem die Möglichkeit die Dimerisierung ohne Ausschnitt der Nukleotide rückgängig zu machen. Der Prozess wird als Reparatur durch Photoreaktivierung (Englisch: "photoreactivation repair") bezeichnet. Das dazu benötigte photoreactivating enzyme (PRE) konnte auch in Pilzen und Pflanzen nachgewiesen werden, jedoch nicht im Menschen. [4]
Medizinische Bedeutung
Die Hautkrankheit Xeroderma pigmentosum wird durch Defekte in verschiedenen Proteinen der Nukleotid-Exzisionreparatur hervorgerufen. Die Zellen können die durch UV-Strahlen hervorgerufenen Läsionen nicht korrigieren.
UV-Strahlen sind der größte Umweltfaktor für die Entstehung von Hautkrebs.
Quellen
- ↑ Goodsell, D. S. The molecular perspective: ultraviolet light and pyrimidine dimers. Oncologist 6, 298-299 (2001).
- ↑ Kim, S. I., Jin, S. G. & Pfeifer, G. P. Formation of cyclobutane pyrimidine dimers at dipyrimidines containing 5-hydroxymethylcytosine. Photochem Photobiol Sci 12, 1409-1415, doi:10.1039/c3pp50037c (2013).
- ↑ Goodsell, D. S. The molecular perspective: ultraviolet light and pyrimidine dimers. Oncologist 6, 298-299 (2001).
- ↑ Klug, W. S. Concepts of genetics. 10th edn, (Pearson Education, 2012). Seite 384-388