Replikation
Synonym: Reduplikation
Englisch: replication
Definition
Die Replikation bezeichnet die Vervielfältigung der Erbinformation nach einem semikonservativen Mechanismus. Das Ergebnis der Replikation sind zwei identische DNA-Moleküle, im Fall einiger Viren zwei RNA-Moleküle.
Basismechanismus
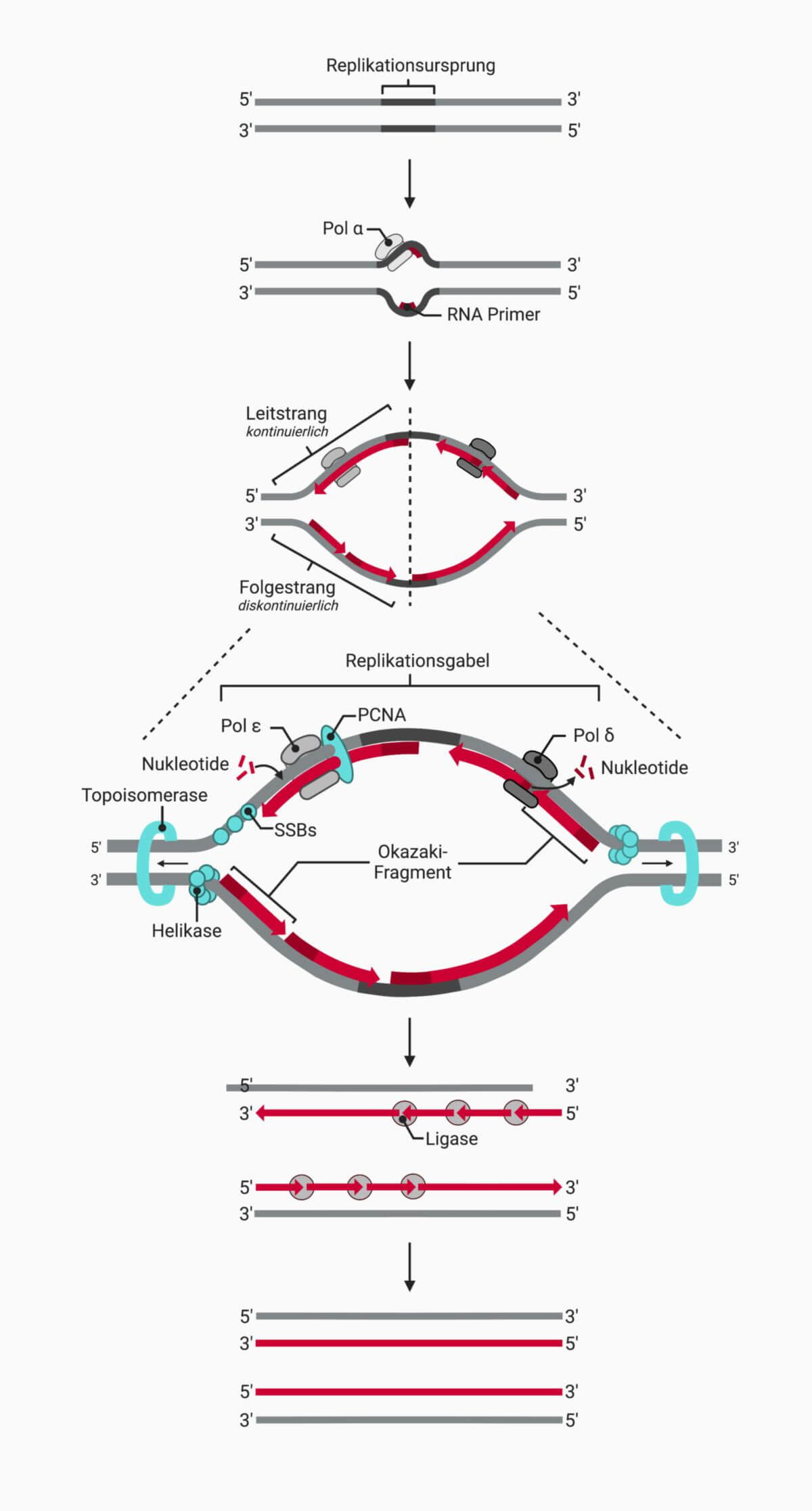
Obwohl die Replikation der DNA in Prokaryoten und Eukaryoten aufgrund der unterschiedlichen Komplexität der Genome einige Unterschiede besitzt, lässt sich doch ein genereller Mechanismus beschreiben:
- Die Replikation startet an definierten Stellen, die als Replikationsursprünge bezeichnet werden.
- Eine Helikase trennt die DNA-Doppelstränge voneinander. Es kommt zur Ausbildung einer Replikationsgabel. Einzelstrang-bindende Proteine (SSBs) stabilisieren die getrennten Stränge.
- Die Doppelhelix-Struktur der DNA erfordert eine Entspiralisierung durch Topoisomerasen im Bereich der Replikation, da sonst die mechanische Spannung auf der DNA steigt und die Helikase die Stränge nicht mehr trennen kann.
- Eine Primase synthetisiert einen RNA-Primer, da DNA-Polymerasen die DNA-Synthese nur an einem freien 3'-OH Ende starten können.
- Eine replikative DNA-Polymerase mit hoher Prozessivität beginnt mit der DNA-Synthese.
- Der strikte 5'→3'-Mechanismus der DNA-Polymerasen führt dazu, dass die DNA-Synthese nur auf einem Strang, dem Leitstrang, kontinuierlich erfolgt. Auf dem anderen Strang (Folgestrang) entstehen beim Fortschreiten der Replikationsgabel immer neue Lücken, da die DNA-Polymerase in entgegengesetzter Richtung synthetisiert. Die DNA-Synthese muss daher regelmäßig erneut durch die Primase initiiert werden. Die Lücken werden dann durch kurze Abschnitte gefüllt, die als Okazaki-Fragmente bezeichnet werden.
- Die RNA-Primer werden aus der DNA entfernt und das Rückgrat der DNA durch eine Ligase verschlossen.
Die Enzyme der Replikation sind durch verschiedene Bindungen miteinander vernetzt und hochgradig koordiniert. Aufgrund dieser Eigenschaften werden sie auch oft als eine molekulare Maschine charakterisiert, die als Replisom bezeichnet wird.
Eukaryoten
Initiation
Die eukaryotische Replikation wird an den Replikationsursprüngen (Englisch: "origins of replication") initiiert. Diese werden durch den Origin recognition complex (ORC) erkannt und gebunden. Eukaryoten besitzen viel komplexere Genome als Prokaryoten. Dies erfordert, dass die Replikation gleichzeitig von vielen Stellen gestartet wird. Die Anzahl der Ursprünge nimmt mit der Komplexität des Organismus zu. Die menschliche DNA besitzt zehntausende Replikationsursprünge, die aber wahrscheinlich nicht alle aktiviert sind. Die Schwierigkeit einen Ursprung zu identifizieren, besteht darin, dass sie nicht durch eine konservierte DNA-Sequenz gekennzeichnet sind, sondern durch eine Struktur, die durch ORC erkannt wird. Diese Bindung rekrutiert verschiedene Proteine, unter anderem die Helikase MCM, die zusammen einen präreplikativen Komplex bilden.
Die Bindung dieser Proteine lizensiert einen Replikationsursprung für die DNA-Synthese, daher werden sie auch als Lizensierungsfaktoren bezeichnet. Sie stellen sicher, dass die Synthese nur einmal pro Zellzyklus und Ursprung gestartet wird, um den kontrollierten und fehlerfreien Ablauf der Replikation zu gewährleisten. Die Bildung des präreplikativen Komplex findet in der G1-Phase des Zellzyklus statt.
Die erfolgreiche Rekrutierung aller Faktoren führt zur Aktivierung von Cyclin-abhängigen Kinasen und zur Trennung der DNA-Stränge (origin firing) durch die Helikase. Die Replikation erfolgt bidirektional, dass heißt sie startet in beide Richtung vom Replikationsursprung. Dementsprechend werden auch zwei Helikasen in entgegengesetzter Orientierung auf den Startpunkt geladen.
Elongation
Untereinheiten der DNA-Polymerase α fungieren als Primasen und synthetisieren kurze RNA-Primer, die sie mit ca. 20 bis 30 DNA-Nukleotiden verlängern. Da die DNA-Polymerase α nur eine geringe Prozessivität und eine hohe Fehlerrate besitzt, wird sie durch eine der replikativen Polymerasen ersetzt. Der Beladung der DNA mit den Polymerasen erfolgt durch PCNA. Die Replikation in Eukaryoten wird durch die DNA-Polymerasen δ und ε durchgeführt. Die Polymerase δ wird durch PCNA auf den Folgestrang geladen und Polymerase ε auf den Leitstrang.
Die Polymerasen synthetisieren nun den komplementären Strang. Im Falle des Folgestrangs erfolgt dies diskontinuierlich, wodurch die Okazaki-Fragmente entstehen. Die Endonuklease FEN1 entfernt den nun seitlich abstehend RNA-Primer und die DNA-Ligase I verbindet die Fragmente.
Termination
Die DNA-Replikation endet, sobald zwei Replikationsgabeln aufeinandertreffen. Da die Genome der Eukaryoten aus linearen DNA-Molekülen bestehen, ergibt sich an den Enden das Problem, dass der Folgestrang nicht bis zum Ende synthetisiert werden kann. Sobald der letzte RNA-Primer eingesetzt ist, kann dieser nicht mehr durch DNA ersetzt werden, wodurch die DNA-Enden mit jeder Zellteilung verkürzt würden. Um dies zu verhindern besitzen die eukaryotischen Chromosomen an diesen Stellen eine lange repetitive Sequenz, die als Telomer bezeichnet wird. Diese werden ohne Matrize durch das Enzym Telomerase synthetisiert.
Prokaryoten
Die bakterielle DNA ist deutlich kleiner, zirkulär und weniger Komplex. Das Genom von Escherichia coli besteht beispielsweise nur aus 4,6 Millionen Basenpaaren, die ca. 4.000 Gene enthalten. Im folgenden wird der Mechanismus der Replikation in Prokaryoten anhand von Escherichia coli erläutert:
Initiation
Die prokaryotische Replikation started von einem einzelnen klar charakterisierten Ursprung, der als OriC bezeichnet wird. OriC besteht aus Wiederholungen einer 13 bp langen Sequenz und einer 9 bp langen Sequenz die DnaA-Box heißt. Diese Boxen werden durch das Protein DnaA erkannt und gebunden. DnaA kann unter ATP-Verbrauch die Trennung der Stränge initiieren. DnaA bindet nun an die Einzelstränge wodurch die Helikase DnaB durch den Helikase-Loader DnaC rekrutiert wird.
DnaA wird nur für die initiale Öffnung der DNA benötigt ist, später während der DNA-Synthese nicht mehr. Der Replikationsgabel läuft die Topoisomerase Gyrase vorraus, welche die DNA entdrillt.
Elongation
Die DNA-Synthese der Prokaryoten ist der eukaryotischen sehr ähnlich. Sie verläuft bidirektional in beide Richtungen. Während die Verlängerung der Nukleotidkette auf dem Leitstrang durch die DNA-Polymerase III kontinuerlich verläuft, muss sie auf dem Folgestrang ca. alle 1.000 bp durch die prokaryotische Primase erneut gestartet werden. Die Elongation dieser Okazaki-Fragmente erfolgt hier jedoch durch dieselbe Polymerase des Leitstranges. Der RNA-Primer wird hier durch die Polymerase I verdrängt und durch DNA ausgetauscht. Eine DNA-Ligase verschließt die Lücke.
Termination
Durch die zirkuläre Struktur der bakteriellen DNA, treffen die Replikationsgabeln am Ende DNA-Synthese wieder aufeinander. Dies geschieht aber nicht an einer zufälligen Region, sondern ist durch eine Terminator-Sequenz gekennzeichnet, die durch das Protein TER gebunden wird und dadurch die Replikationsgabeln aufhält.
Virusreplikation
In der Virologie wird die Virusreplikation, also die Vermehrung eines Virus unter Zuhilfenahme des Wirtsstoffwechsels, als Replikation bezeichnet. DNA-Viren sind meist komplett auf die Replikationsmaschinerie der Wirtszelle angewiesen. Einige besitzen jedoch Gene für eine eigene DNA-Polymerase (z.B. T4-Polymerase). RNA-Viren besitzen immer eine eigene spezifische RNA-abhängige RNA-Polymerase (RdRP) und eine reverse Transkriptase, um die ihr Erbgut in das Wirtsgenom integrieren zu können.
siehe auch: Virusreplikation
Literatur
- Anthony J.F. Griffiths: Introduction to Genetic Analysis. W.H. Freeman and Company, 2005, Seite 246-249.
- Klug, W. S. Concepts of genetics. 10th edn. Pearson Education, 2012, Seite 269-286.