Single-Strand Annealing
Definition
Single-Strand Annealing, kurz SSA, ist ein zellulärer Reparaturmechanismus für Doppelstrangbrüche. Er erfordert homologe Sequenz-Wiederholungen (Tandem Repeats) auf beiden Seiten des Bruchs. SSA führt immer zum Verlust aller Sequenzen die sich zwischen Bruch und den beiden Tandem Repeats befinden und besitzt hierdurch ein großes Mutationspotential.
Mechanismus
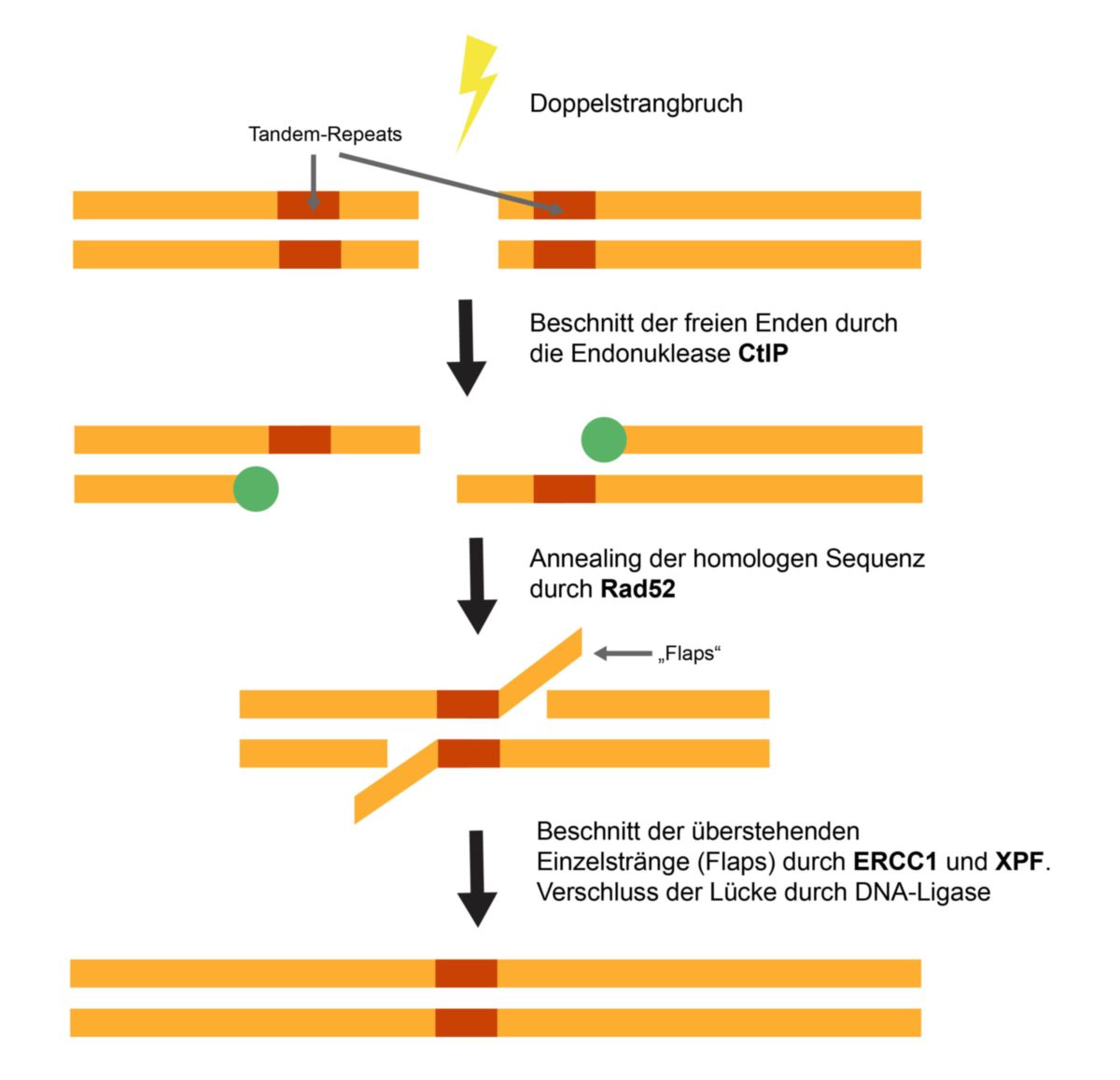
- Doppelstrangbruch:
Ein Doppelstrangbruch zertrennt beide DNA-Stränge. - Erzeugung der 3'-Überhänge (end resection):
Die Endonuklease CtIP beginnt die Stränge zu beschneiden, bis sie auf die homologen Sequenzen der Tandem Repeats trifft. Es entstehen dadurch einzelsträngige 3'-Überhänge. - Annealing der Überhänge:
Das DNA-bindende Protein Rad52 vermittelt nun das Annealing der homologen Sequenzen. - Entfernung der überstehenden Klappen:
Durch das Annealing stehen die 3'-Überhänge von der DNA klappenartig ab ("flaps"). Diese werden durch die Nukleasenfunktion des ERCC1-XPF-Komplexes, abgeschnitten. - Verschluss der Lücken:
Die Lücken werden durch DNA-Polymerasen und DNA-Ligasen wieder verschlossen.
Abhängigkeit vom Zellzyklus
Die Reparatur mittels SSA ist nur in einem kurzen Bereich des Zellzyklus präsent: In der frühen S-Phase, noch bevor die Replikation stattgefunden hat. SSA erfordert beschnittene Strangenden. Die dafür benötigte Endonuklease CtIP ist jedoch nur in der S-Phase bis G2-Phase aktiv. Das Vorliegen eines Schwesterchromatids ist keine Voraussetzung für den Beschnitt der Enden. SSA kann zwar auch in der G2-Phase, beim Vorliegen eines Schwesterchromoatids durchgeführt werden, jedoch führt die Zelle hier die Reparatur mittels homologer Rekombination deutlich häufiger durch. Ein wichtiger Faktor der die Reparatur mittels HR vermittelt, ist das Protein TP53BP1.
Spezifität und Fehleranfälligkeit
Befinden sich mehrere Tandem Repeats nebeneinander, kann theoretisch jedes als Ausgangspunkt der Reparatur verwendet werden. Es werden jedoch die Repeats in nächster Nähe zum Bruch bevorzugt. Die verwendeten Repeats können jedoch sehr weit voneinander entfernt sein. In Saccharomyces cerevisiae wurde gezeigt, dass die Repeats über 25.000 bp voneinander entfernt sein können. Dies hat zur Folge, dass auch sehr große Sequenzbereiche deletiert werden können. SSA besitzt daher ein enormes Mutationspotential.[1]
Verwandtschaft zu Microhomology-mediated end Joining
Der Mechanismus des Single-Strand Annealings zeigt Ähnlichkeiten zum Microhomology-mediated end joining (MMEJ), das ebenfalls kurze Homologien verwendet, um Doppelstrangbrüche zu reparieren. Es wird daher diskutiert, ob es sich beim SSA nicht um eine Unterform des MMEJ handelt. Obwohl einige Proteine in beiden Mechanismen vorkommen, beispielsweise XPF–ERCC1, EXO1 und Srs2, tendieren die meisten Studien mittlerweile dazu, sie als eigenständige Prozesse zu betrachten.[2]
Quellen
- ↑ Bhargava, R., Onyango, D. O. & Stark, J. M. Regulation of Single-Strand Annealing and its Role in Genome Maintenance. Trends Genet 32, 566-575, doi:10.1016/j.tig.2016.06.007 (2016).
- ↑ McVey, M. & Lee, S. E. MMEJ repair of double-strand breaks (director's cut): deleted sequences and alternative endings. Trends Genet 24, 529-538, doi:10.1016/j.tig.2008.08.007 (2008).