Proteinstruktur
Synonym: Proteinkonformation
Englisch: protein structure
Definition
Unter Proteinstruktur versteht man in der Biochemie verschiedene Strukturebenen von Proteinen bzw. Peptiden. Man teilt diese Strukturen hierarchisch auf in eine Primärstruktur (Aminosäuresequenz), Sekundärstruktur, Tertiärstruktur und Quartärstruktur. Wird ein Protein in Bezug auf die räumliche Anordnung erwähnt, spricht man auch oft vom Begriff Proteinkonformation. Daraus resultiert auch der Begriff Konformationsänderung, der eine Änderung der räumlichen Proteinstruktur bedeutet.
Hintergrund
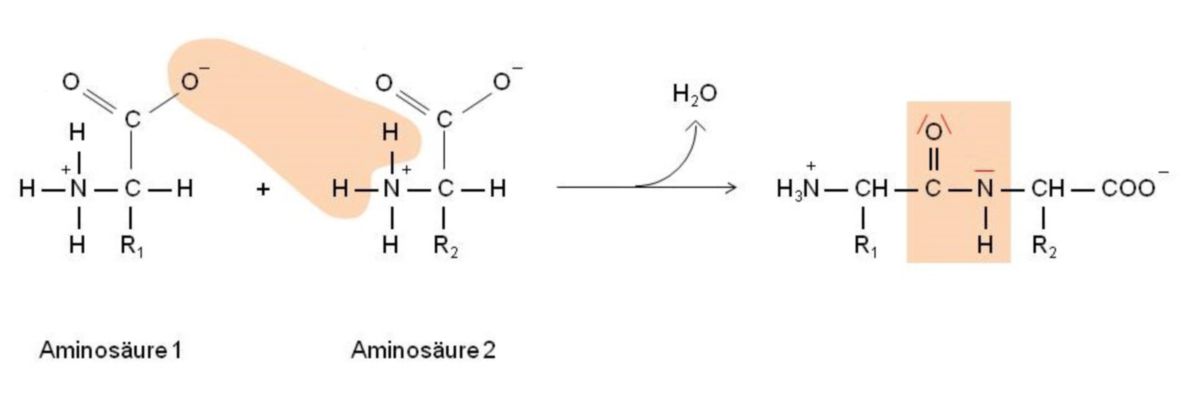
Grundlage für die Anordnung von Proteinen ist die Peptidbindung. Sie ist die Bindungsart, die Aminosäuren in der Regel immer auf die gleiche Weise miteinander verbindet. In Zellen werden Peptidbindungen normalerweise unter Vermittlung von Ribosomen gebildet. Letztendlich entspricht eine Peptidbindung einer Verbindung zwischen der Carboxylgruppe einer Aminosäure und der Aminogruppe einer anderen Aminosäure unter Abspaltung von Wasser (Hydrolyse).
In der Peptidbindung ist eine C=O-Gruppe nur durch eine Einfachbindung mit einer NH-Gruppe verbunden ist. Das Stickstoffatom trägt ein freies Elektronenpaar, das unter dem elektronenziehenden Einfluss des Sauerstoffatoms steht (auf Grund der hohen Elektronegativität des Sauerstoffs). Somit zieht das Sauerstoff das freie Elektronenpaar zu einem gewissen Teil in die Bindung zwischen dem Stickstoff- und dem Kohlenstoffatom hinein, so dass die Peptidbindung einen partiellen Doppelbindungscharakter erhält. Die enstandenen Doppelbindungscharakter bewirkt eine Aufhebung der freien Drehbarkeit der C=O-Gruppe und der NH-Gruppe.
Das Sauerstoff- und Wasserstoffatom der Peptidbindungen spielen im Zusammenhang mit der Strukturbildung aller Peptide und Proteine eine entscheidende Rolle. Es ist möglich, dass sich zwei Aminosäuren so aneinander anlagern, dass sich sämtliche Peptidbindungen der beiden Aminosäurenketten direkt gegenüberliegen. Die Wasserstoffatome der Peptidbindungen sind im Vergleich zu den Sauerstoffatomen der nun direkt gegenüberliegenden Peptidbindungen vergleichsweise positiv polarisiert. Resultat ist die Ausbildung sog. Wasserstoffbrücken, die durch die beiden Aminosäureketten miteinander verbunden werden.
| Ausgebildete Wasserstoffbrücken tragen einen wesentlichen Anteil zur Bildung von Proteinstrukturen bei. |
Allgemeines
Unter der nativen Struktur eines Proteins versteht man die definierte dreidimensionale Struktur, in der das Protein seine physiologische Funktion ausübt. Aminosäuren, die in kleinen Peptiden enthalten sind, können untereinander nur wenige Wechselwirkungen eingehen. Sie bilden deshalb im Gegensatz zu den Proteinen in der Regel keine stabilen Strukturen aus. Solange ein vorliegendes Protein seine native Struktur noch nicht eingenommen hat, liegt es in einer nicht-nativen Struktur vor. Verliert ein Protein nachträglich seine native Struktur, wird es in diesem Moment gerade denaturiert.
In den Zellen können denaturierte Proteine nur teilweise wieder in den nativen Zustand zurückversetzt werden. Meist ist eine abgelaufene Denaturierung irreversibel. Die somit funktionell unbrauchbaren, denaturierten Proteine werden anschließend von Proteasen hydrolysiert und aus den daraus freigesetzten Aminosäuren werden wieder neue Polypeptide synthetisiert.
Der Begriff Proteindomäne bezeichnet einen größeren Teil einer Aminosäurekette, der unabhängig von den anderen Proteinanteilen eine eigene dreidimensionale Struktur ausbildet. In der Regel bilden derartige Proteindomänen im menschlichen Körper auch funktionelle Einheiten des Proteins.
Einteilung
Primärstruktur
Der Begriff Primärstruktur in Bezug auf ein Protein beschreibt die Aminosäuresequenz. Wird ein Protein also denaturiert (beispielsweise durch Verbrennungen oder Verbrühungen der Haut), geht zwar seine native Struktur verloren, die Primärstruktur aber bleibt erhalten. Die Primärstruktur allein reicht nicht für die Erhaltung der Proteinfunktion aus. Die native Struktur ist hierfür erforderlich, die durch die Sekundär-, Tertiär- und Quartärfunktion bestimmt wird.
Bildet man eine Aminosäuresequenz ab, stellt man die freie Aminogruppe der ersten Aminosäure konventionsgemäß auf die linke Seite, die freie Carboxylgruppe der letzten Aminosäure rechts dar. Dementsprechend unterscheidet man auch zwischen einem N-terminalen und einem C-terminalen Ende. Bezogen auf den Menschen, beginnt mit der N-terminalen Aminosäure am Ribosom die Proteinbiosynthese.
Sekundärstruktur
Regelmäßige Strukturen innerhalb von Polypeptiden, die sich aufgrund von ausgebildeten Wasserstoffbrücken zwischen Peptidbindungen aneinanderheften, werden als Sekundärstruktur bezeichnet. Da Aminosäuresequenzen verschiedene Abschnitte aufweisen, zeigen diese in der Regel unterschiedliche Sekundärstrukturen.
Die Aminosäurereste der verschiedenen Aminosäuren eines Proteins können zwar die Ausbildung einer bestimmten Sekundärstruktur wesentlich begünstigen, sind jedoch an der Ausbildung der entscheidenden Wasserstoffbrücken der Sekundärstruktur nicht beteiligt.
Untereinteilung
Da jedes Protein mehrere Sekundärstrukturelemente enthält, lassen sich diese wiederum unterteilen, in:
- α-Helix: Es handelt sich hierbei um eine rechtsgängige Schraube, bei der jeweils 3,6 Aminosäuren eine Windung beisteuern. Außerdem hat jede Windung eine Ganghöhe von 0,54 nm und wird ausschließlich durch intramolekulare Wasserstoffbrücken stabilisiert.
- β-Faltblatt: Hier lagern sich Aminosäurenketten weitgehend in gestreckter Konformation nebeneinander und bilden so ein Beta-Faltblatt aus. Die einzelnen Faltblattstrukturen werden durch Wasserstoffbrücken stabilisiert, die sich zwischen zwei parallel oder auch antiparallel liegenen Aminosäureketten ausbilden.
- U-förmige Verbindungsstücke (engl. loop bzw. turn, deutsch: Schleife): Solche Schleifen sind U-förmige Abschnitte der Aminosäurekette, die die Alpha-Helices und Beta-Faltblattstrukturen eines Proteins miteinander verbinden.
Tertiärstruktur
Mithilfe der Tertiärstruktur wird die räumliche Struktur einer kompletten Aminosäurekette beschrieben, einschließlich der Anordnung sämtlicher Aminosäurereste. Die Tertiärstruktur eines Proteins wird nicht durch die Wasserstoffbrückenbindungen, sondern durch die Wechselwirkung der Aminosäurereste stabilisiert. Diese Kräfte (Wechselwirkungen), die bei der Stabilisierung einer Tertiärstruktur beteiligt sind, weisen eine gleich große Vielfalt auf, wie die funktionellen Gruppen der verschiedenen Aminosäuren. So können in unterschiedlichem Ausmaß beteiligt sein:
Quartärstruktur
Durch die Quartärstruktur wird die Zahl und die Anordnung der verschiedenen Aminosäureketten in einem Proteinkomplex beschrieben. Somit ist eine Quartärstruktur nur dann gegeben, wenn sich mehrere Aminosäureketten zu einem Komplex zusammenlagern.
Ausbildung einer Raumstruktur
Der Prozess, der zu einer dreidimensionalen Raumerfüllung eines Proteins führt, erfolgt teilweise spontan (während der Translation), teilweise aber auch durch die Mitwirkung von Enzymen oder Chaperonen. Liganden beeinflussen ebenfalls die Proteinstruktur, so dass manche Proteine - je nach Komplexierung mit Cofaktoren oder Substraten - verschiedene Strukturen einnehmen können (Konformationsänderung). Die Fähigkeit, die Raumstruktur zu verändern, ist für viele Enzymaktivitäten wichtig.
Strukturbestimmung
Um herauszufinden, welche räumliche Proteinstruktur gegeben ist, werden verschiedenste experimentelle Methoden zur Hand genommen, u.a.:
- Kristallstrukturanalyse: Erstellt meist mittels Röntgenstrahlen das Beugungsbild eines Proteinkristalls, woraus man dessen dreidimensionale Struktur errechnen kann
- NMR-Spektroskopie: Ermöglicht das Ermitteln der Struktur eines Proteins in einer Lösung, was den physiologischen Bedingungen des Portein eher entspricht. Leider ergibt sich hierbei keine eindeutige Struktur, da sich die Atome des Proteins in solch einem Zustand bewegen. Um nun zu einer eindeutig erkennbaren Struktur zu gelangen, wird meist über die abgebildeten Strukturen ermittelt.
Die Proteinstruktur ist von physikochemischen Randbedingungen wie pH-Wert, Temperatur, Salzgehalt, Gegenwart anderer Proteine, etc. abhängig. Über eine Native-PAGE, eine Größenausschlusschromatographie oder über eine isopyknische Zentrifugation kann der Stokes-Radius eines nativen Proteins ermittelt werden. Beide Methoden könne mit einer Quervernetzung oder mit einem Alaninscan kombiniert werden.
Strukturvorhersage
Gibt es bereits Proteine, die eine ähnliche Sequenz und eine bekannte Struktur aufweisen, kann man bei der Vorhersage räumlicher Proteinstrukturen (Proteinstrukturvorhersage) gute Ergebnisse erzielen. Ermöglicht wird dies durch das sogenannte homology modelling. Hierbei wird die neue Sequenz auf die Sequenz, dessen Struktur schon bekannt ist, abgebildet und damit in die Struktur quasi "eingepasst, eingefügt". Dieses Verfahren ähnelt stark dem Sequenzalignment.
Klinik
Liegt eine Störung in der Bildung einer funktionsfähigen Raumstruktur vor, so werden diese als Proteinfaltungserkrankungen bezeichnet. Ein Beispiel dafür ist Chorea Huntington. Auch Prionkrankheiten wie BSE oder Morbus Alzheimer werden durch Fehlbildungen der Proteinstruktur ausgelöst.