DNA Damage Response
Deutsch: DNA-Schadensantwort
Definition
Die DNA Damage Response, kurz DDR, ist der englische Fachbegriff für die Gesamtheit aller zellulären Mechanismen, die nach einem DNA-Schaden aktiviert werden. Er umfasst nicht nur die unmittelbare Reparatur des Schadens, sondern auch Signalwege, die das Überleben der Zelle kontrollieren.
Gelegentlich wird auch in der deutschsprachigen Literatur die Übersetzung DNA-Schadensantwort verwendet, jedoch ist der englische Begriff geläufiger.
Hintergrund
In jeder menschlichen Zelle kommt es pro Tag zu etwa 10.000-100.000 Beschädigungen der DNA. Die Art dieser Schäden reicht von der Oxidation einer DNA-Base, bis zum kompletten Bruch beider Stränge. Die menschliche Zelle besitzt eine Vielzahl von Reparaturmechanismen, um die Schäden zu korrigieren. Parallel wird eine globale Reaktion initiiert, um die Integrität des Genoms sicherzustellen. Bei massiven Beschädigungen des Genoms wird die Zelle an der Replikation gehindert.[1]
Ein Merkmal der DNA Damage Response ist die starke Redundanz der Mechanismen. Ist ein Protein durch eine Mutation nicht mehr Funktionsfähig, kann dies meist durch ein anderes Protein kompensiert werden.
Übersicht
Ablauf
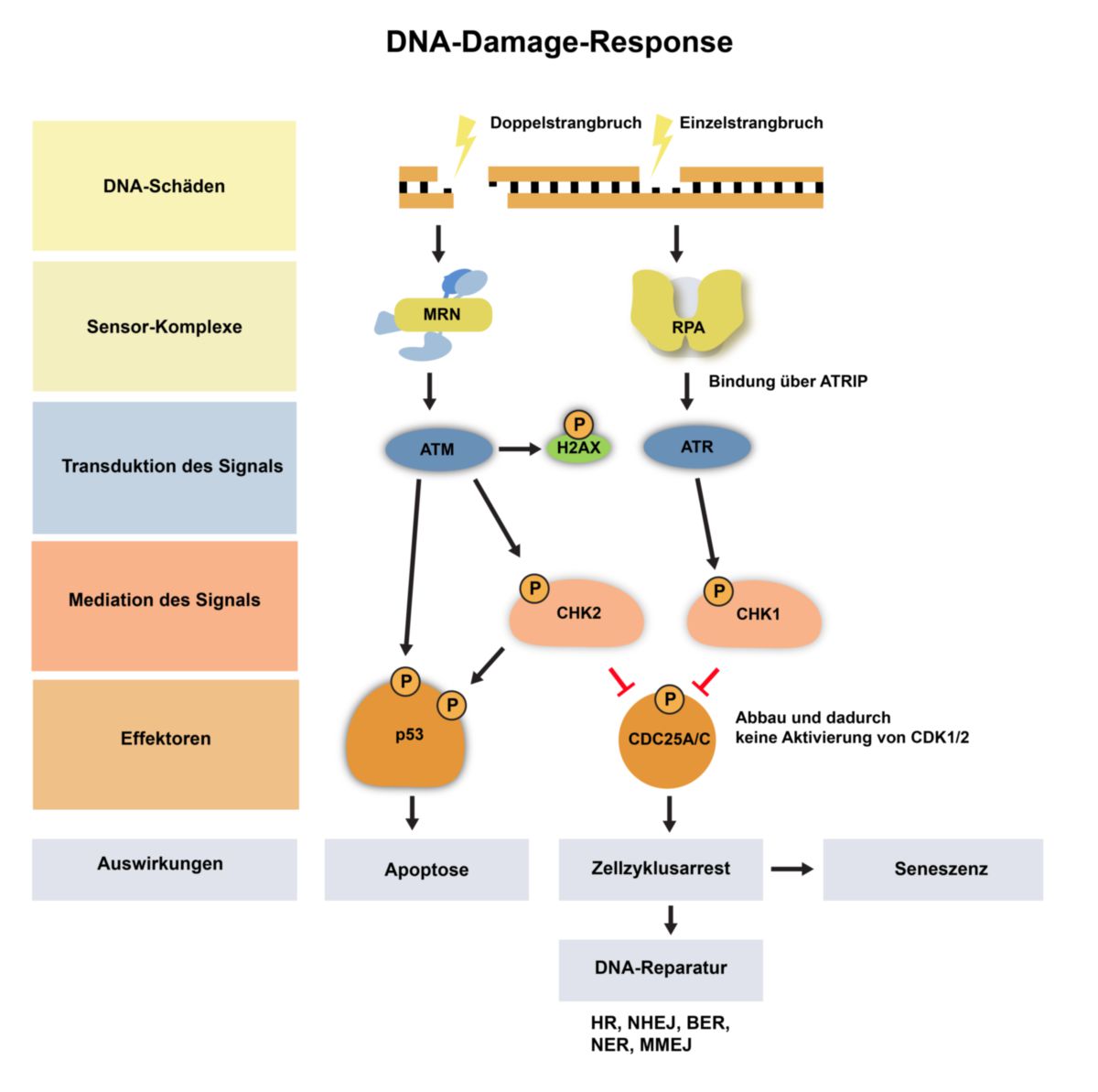
Die Proteine und Mechanismen können in fünf Bereiche unterteilt werden:
- Detektion eines DNA-Schadens
- Transduktion des Signals
- Mediation des Signals
- Effektoren
- Reaktion auf den Schaden
Konsequenzen
- Die Beschädigung konnte repariert werden. Die Zelle kann normal im Zellzyklus fortfahren.
- Die Beschädigung konnte nicht hinreichend repariert werden, die Zelle geht aber nicht in Apoptose und wird an weiterer Zellteilung gehindert: Zelluläre Seneszenz
- Die Beschädigungen können nicht mehr durch Zelle repariert werden: Apoptose
Mechanismus
Detektion
Doppelstrangbrüche werden innerhalb weniger Sekunden durch den MRN-Komplex detektiert, der an freie Strangenden bindet. Das Protein RPA bindet hingegen an freie einzelsträngige DNA, wie sie beispielsweise bei einer blockierten Replikationsgabel entsteht.
Transduktion des Signals
Die Aktivierung der eigentlichen Antwort erfolgt durch zwei apikale Kinasen: ATM und ATR. Beide sind sehr große Proteine mit einer vergleichbaren Domänenstruktur. Sie besitzen eine lange N-terminale HEAT-Domäne, die zahlreiche Protein-Protein Interaktionen ermöglicht und eine C-terminale Kinasendomäne.
ATM und ATR werden ebenfalls innerhalb weniger Sekunden zur beschädigten Stelle rekrutiert. ATM bindet an den MRN-Komplex und ATR bindet über das Protein ATRIP an die mit RPA bedeckten Einzelstrangbrüche. Diese Bindung führt zur Aktivierung der Kinasen. ATR und ATM haben viele gemeinsame Substrate, jedoch gilt ATM als globaler Regulator der DDR durch die große Anzahl der Proteine, die durch ATM phosphoryliert werden. Die Histonvariante H2AX wird durch ATM aktiviert und agiert einerseits als Signalverstärker, verändert andererseits die Zugänglichkeit des Chromatins für Reparaturfaktoren.
Zusätzlich können Doppelstrangbrüche auch durch Ku70/Ku80 detektiert werden. Hierdurch wird die Kinase DNA-PKcs rekrutiert. Das daraus entstehende Holoenzym DNA-PK führt jedoch zu einer deutlich geringeren Aktivierung anderer Proteine. DNA-PK ist vorrangig in die Vermittlung der Reparatur durch die Nicht-homologe Endverknüpfung involviert.[2]
Aktivierung von Mediatoren
Die Mediatorproteine sind für die Feinregulierung der DNA Damage Response zuständig. Eine zentrale Bedeutung kommt hier den Checkpointkinasen 1 und 2 zu. CHK1 wird durch ATR phosphoryliert und ist ein wichtiger Regulator der Replikation, indem es den Zellzyklus anhält und der Zelle Zeit für die Aufhebung blockierter Replikationsgabel verschafft. CHK2 rekrutiert Reparaturproteine wie BRCA1 und kontrolliert die Zellzyklus-Kontrollpunkte analog zu CHK1.
Effektoren
Die Checkpointkinasen verhindern das Fortschreiten im Zellzyklus, indem die Cyclin-abhängigen-Kinasen nicht mehr aktiviert werden können. Dies geschieht durch Inhibierung der Phosphatasen Cdc25A und Cdc25C, die eine hemmende Phosphatgruppe entfernen können. Die Zellen werden dadurch in der S-Phase oder G2-Phase arretiert.
ATM und CHK2 phosphorylieren an verschieden Aminosäuren den zentralen Tumorsuppressor p53. Dieser liegt in unbeschädigten Zellen inaktiviert an Mdm2 gebunden vor. Die Phosphorylierungen führen zur Ablösung und stabilisieren p53 in der Zelle.
Auswirkungen
Doppelstrangbrüche können in der Regel durch homologe Rekombination mit hoher Effizienz fehlerfrei repariert werden. Die Zelle kann die Replikation beenden und mit der normalen Proliferation fortfahren. Massive Beschädigungen können die Kapazitäten der Reparaturmechanismen jedoch erschöpfen. Dies führt zu einer Akkumulation von p53, wodurch weitere pro-apoptotische Faktoren aktiviert werden und der Zelltod eingeleitet wird.
Alternativ wird die Zellseneszens ausgelöst. Die Zellen können die Schäden nicht mehr korrigieren und werden an der Zellteilung gehindert. Es konnte gezeigt werden, dass hier die Reparatur-Foci bestehen bleiben.[3]
Klinische Bedeutung
Die DNA Damage Response ist in Krebszellen fast immer dereguliert, wodurch ihre unbegrenzte Proliferation ermöglicht wird. Daher ist genomische Instabilität ein typisches Merkmal von Krebs.
Für die rapide Proliferation ist es jedoch erforderlich, dass DNA-Schäden, welche die Replikation behindern, entfernt werden. Diese Zellen sind dann oft von wenigen noch intakten Reparaturmechanismen abhängig. Werden diese durch Chemotherapeutika inhibiert, können sie im Vergleich zu gesunden Zellen nicht auf andere Mechanismen ausweichen und gehen in Apoptose. Die starke Redundanz der DNA Damage Response macht es jedoch erforderlich, das mehrere Signalwege parallel blockiert werden.[4] Bekannte Inhibitoren und Ziele sind:
| Protein | Inhibitor |
|---|---|
| ATM | KU-55933 |
| ATM | KU-60019 |
| ATR | NU6027 |
| ATR | AZ20 |
| CHK1/CHK2 | XL844 |
| CHK1/CHK2 | AZD7762 |
| CHK1 | PF-477736 |
Beispiele für Erkrankungen, die durch Mutationen in Genen der DNA Damage Response entstehen:
| Mutation | Erkrankung |
|---|---|
| p53 | Li-Fraumeni-Syndrom |
| CHK2 | Mammakarzinom |
| ATM | Ataxia telangiectasia |
| MRE11 | Ataxia telangiectasia-like disorder |
| NBS1 | Nijmegen-Breakage-Syndrom |
| BRCA1 | Mammakarzinom |
Weiterführende Links
Komplexere Darstellungen der DNA Damage Response:
siehe auch: DNA-Schaden, DNA-Reparatur
Quellen
- ↑ Jackson, Stephen P., and Jiri Bartek. “The DNA-Damage Response in Human Biology and Disease.” Nature 461.7267 (2009): 1071–1078. PMC. Web. 16 Feb. 2018.
- ↑ Hosoya, N. & Miyagawa, K. Targeting DNA damage response in cancer therapy. Cancer Sci 105, 370-388, doi:10.1111/cas.12366 (2014).
- ↑ Galbiati, Alessandro, Christian Beauséjour, and Fabrizio d’ Adda di Fagagna. “A Novel Single‐cell Method Provides Direct Evidence of Persistent DNA Damage in Senescent Cells and Aged Mammalian Tissues.” Aging Cell 16.2 (2017): 422–427. PMC. Web. 16 Feb. 2018.
- ↑ Hosoya, N. & Miyagawa, K. Targeting DNA damage response in cancer therapy. Cancer Sci 105, 370-388, doi:10.1111/cas.12366 (2014).