MRN-Komplex
Synonym: MRE11-RAD50-NBS1-Komplex
Definition
Der MRN-Komplex ist ein Enzymkomplex, der aus den Proteinen MRE11, RAD50 und NBS1 besteht. Dieser Komplex ist ein Sensor für DNA-Doppelstrangbrüche und ist direkt in die Reparatur durch homologe Rekombination und Nicht-homologe Endverknüpfung involviert.
Struktur
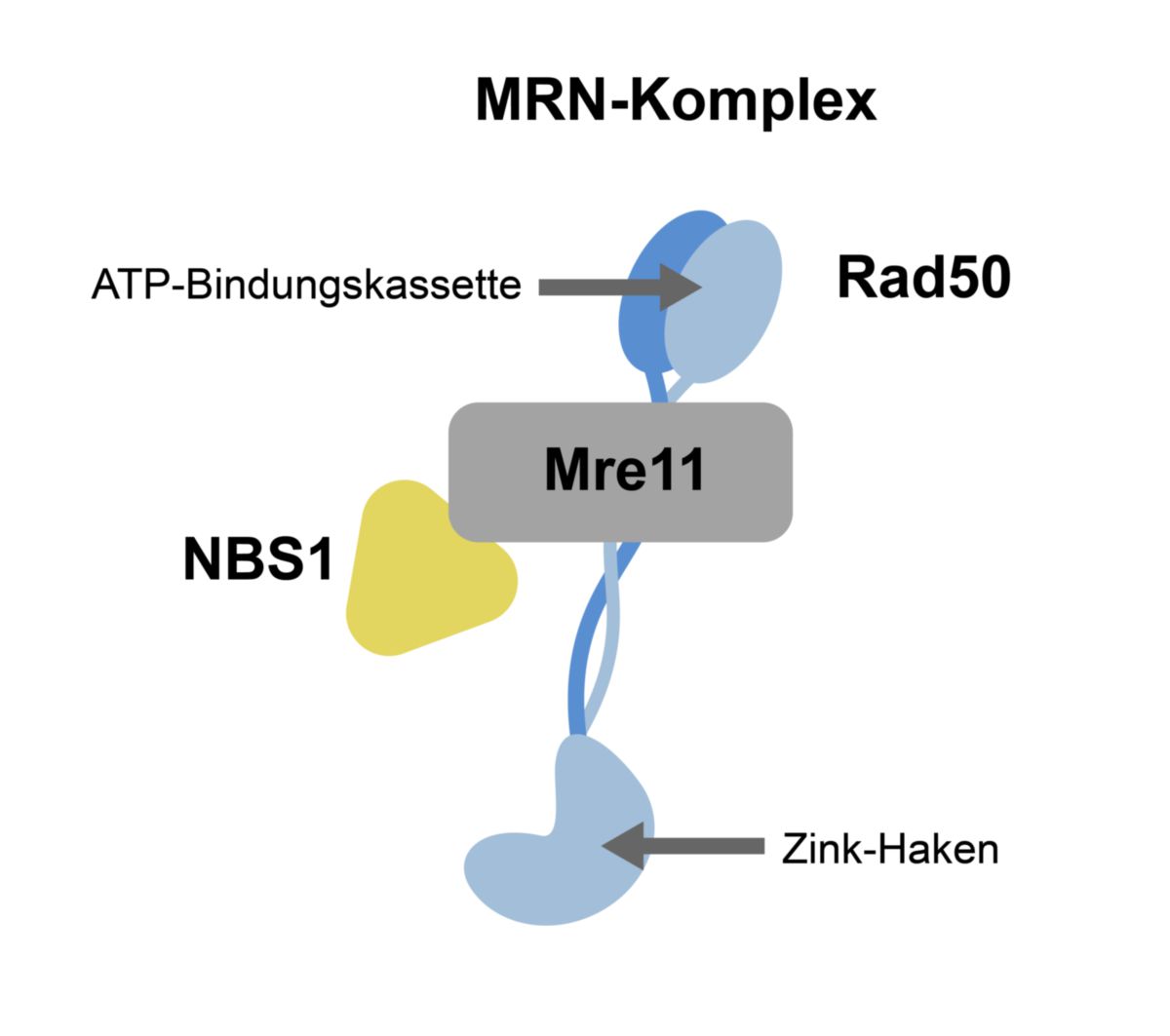
MRN besteht aus drei nicht verwandten Proteinen und ist in unbeschädigten Zellen gleichmäßig im Nukleus verteilt sind.[1]
MRE11
MRE11 besitzt zwei C-terminale DNA-Bindungsdomänen und zeigt Endonukleasen- und Exonukleasenaktivität. Außerhalb des MRN-Komplexes kann MRE11 stabile Dimere bilden.
RAD50
Rad50 bildet eine längliche Coiled-Coil-Struktur aus, wodurch eine zweiteilige ATP-Bindungskassette entsteht. Diese befindet sich nach Bindung an MRE11 in direkter Nähe zu deren DNA-Bindungsdomäne. Am anderen Ende der Coiled-Coil-Struktur befindet sich eine Zink-Haken-Domäne. Durch diesen Zink-Haken können die zwei Enden des Doppelstrangbruchs miteinander verbunden werden.
NBS1
NBS1 rekrutiert Zellzyklus- und DNA-Reparaturproteine zum Doppelstrangbruch. NBS1 bindet durch eine Domäne am C-Terminus an MRE11.
Funktion bei Doppelstrangbrüchen
Detektion
Die zelluläre MRN Konzentration bleibt während des gesamten Zellzyklus konstant im gesamten Nukleus verteilt. Nach einem Doppelstrangbruch bindet MRN innerhalb von 15-30 Sekunden die offenen Strangenden. Zusätzlich werden auch hunderte weitere MRN-Moleküle in die direkte Nähe des Schadens relokalisiert. ATM bindet an NBS1 wodurch ATM monomerisiert und aktiviert wird. Dies initiiert die DNA-Schadensantwort (Englisch: "DNA-damage Response") der Zelle.[2]
Verarbeitung der Strangenden
Die Verarbeitung der Strangenden entscheidet, ob der Doppelstrangbruch mittels Nicht-homologer Endverknüpfung (NHEJ) oder homologer Rekombination (HR) repariert wird. HR benötigt längere einzelsträngige DNA-Abschnitte, damit die HR-spezifischen Faktoren Rad51 und RPA binden können. Dies verhindert wiederum die Bindung der NHEJ-spezifischen Faktoren Ku70/Ku80. Die Beschneidung findet mittels der Exo- und Endonukleasenaktivität von MRE11 statt.[3]
Mutationen
Das Nijmegen-Breakage-Syndrom (NBS) und Ataxia-telangiectasia-like disorder (ATLD), werden durch Mutationen im MRN-Komplex ausgelöst. Beide Krankheitsbilder sind mit genetischer Instabilität und einer stark erhöhten Krebsdisposition assoziiert.[4]
Quellen
- ↑ Lamarche, Brandon J, Nicole I Orazio, and Matthew D Weitzman. “The MRN Complex in Double-Strand Break Repair and Telomere Maintenance.” FEBS letters 584.17 (2010): 3682–3695. PMC. Web. 6 Feb. 2018.
- ↑ Hartlerode, Andrea J. et al. “Recruitment and Activation of the ATM Kinase in the Absence of DNA Damage Sensors.” Nature structural & molecular biology 22.9 (2015): 736–743. PMC. Web. 6 Feb. 2018.
- ↑ Lamarche, Brandon J, Nicole I Orazio, and Matthew D Weitzman. “The MRN Complex in Double-Strand Break Repair and Telomere Maintenance.” FEBS letters 584.17 (2010): 3682–3695. PMC. Web. 6 Feb. 2018.
- ↑ Czornak, K., Chughtai, S. & Chrzanowska, K.H. J Appl Genet (2008) 49: 383. https://doi.org/10.1007/BF03195638