CRISPR/Cas-System
Synonyme: CRISPR/Cas9, CRISPR/Cas-Methode
Definition
Als CRISPR/Cas-System bezeichnet man eine gentechnische Methode, mit der gezielt die DNA-Sequenz im Genom eines Zielorganismus modifiziert werden kann.
Hintergrund
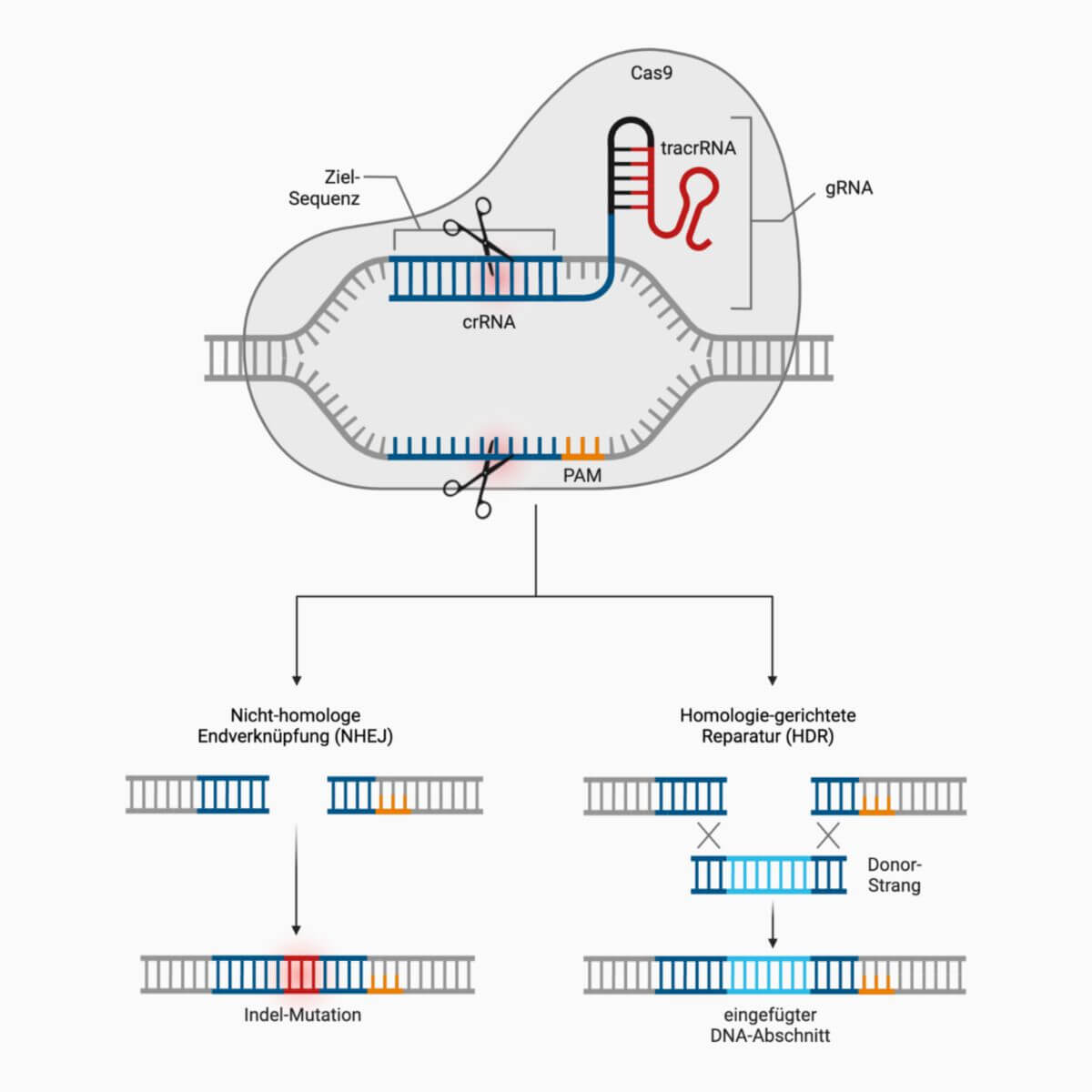
Das CRISPR/Cas-System beruht auf einem bakteriellen Abwehrsystem gegenüber fremder DNA, die z.B. durch eine Phagen-Infektion in die Zelle eingebracht wird. Bakterien besitzen verschiedene dieser sogenannten CRISPR-Systeme, gentechnologisch genutzt wird aktuell (2023) überwiegend der Typ II aus Streptococcus pyogenes. Dieses System produziert kleine RNA-Moleküle (crRNAs), die komplementär zu bestimmten Abschnitten der Fremd-DNA sind. Nach Hybridisierung mit sogenannten tracrRNAs bildet sich ein Ribonukleoprotein aus, das eine Nuklease enthält. Das Ribonukleoprotein wird durch die Sequenz der crRNAs zur Fremd-DNA gelotst und baut sie ab. Dadurch wird die Vermehrung der Phagen verhindert.
Das CRISPR-System Typ II verwendet die Nuklease Cas9, daher wird bei der gentechnisch genutzten Variante häufig auch von CRISPR/Cas9 gesprochen. Ebenso häufig wird auch nur der Begriff CRISPR verwendet. Streng genommen bezeichnet dieses Akronym ("clustered regularly interspaced short palindromic repeats") aber nur einen Abschnitt im CRISPR-Locus, der weitere Komponenten enthält.
Prinzip
Das gentechnisch eingesetzte CRISPR/Cas-System erzeugt spezifisch sogenannte Doppelstrangbrüche in der Ziel-DNA. Dabei durchtrennt die Nuklease die beiden komplementären Stränge. Diese Verletzung der DNA wird durch Reparaturmechanismen behoben. Während dieses Prozesses können gewollte Modifikationen induziert werden.
Voraussetzung
Um das CRISPR/Cas-System in einem Organismus nutzen zu können, müssen zwei Komponenten in diesem über Vektoren exprimiert werden:
- die Nuklease Cas9
- eine sogenannte guide RNA (gRNA)
Guide RNA
Die gRNA ist das Instrument, das die Spezifität des CRISPR/Cas-System bestimmt. Sie ist ein Fusionsprodukt aus crRNA und tracrRNA. Die crRNA besteht hier aus
- einer Sequenz mit 20 Nukleotiden, die komplementär zur Zielsequenz in der DNA ist, die modifiziert werden soll und
- einem Abschnitt, der mit einem Teil der tracrRNA hybridisieren kann.
Die tracrRNA besteht immer aus der gleichen Sequenz, die crRNA wird je nach Ziel-DNA angepasst. Im Vergleich zum bakteriellen System sind die beiden Abschnitte in der gRNA kovalent verbunden. Die gRNA bildet eine Sekundärstruktur mit mehreren Haarnadelstrukturen aus, die für die Interaktion mit der Nuklease notwendig ist.
Ziel-Sequenz und PAM
Theoretisch kann die gRNA so konzipiert werden, dass sie zu jedem Abschnitt in der Ziel-DNA komplementär ist. Damit die Nuklease die DNA schneiden kann, muss die Sequenz aber in unmittelbarer Nähe zu einem sogenannten Protospacer adjacent motif (PAM) liegen. Im bakteriellen System markiert diese Sequenz, welche Abschnitte aus der fremden DNA ausgeschnitten werden.
Für Cas9 lautet das PAM-NGG, d.h. auf ein beliebiges Nukleotid folgen zwei Guanin-Nukleotide. Diese Sequenz muss direkt downstream der Zielsequenz liegen, damit sie von Cas9 erkannt und geschnitten werden kann. In der Folge kann im Genom eines Organismus also jede Sequenz mit der Form N20-NGG mit dem CRISPR/Cas9-System verändert werden.
Reparatur des Doppelstrangbruchs
Der eigentliche Prozess, mit dem die Modifikation der Ziel-DNA hervorgerufen wird, ist die Reparatur des Doppelstrangbruchs, der durch die Aktivität von Cas9 entstanden ist. In Säugerzellen geschieht dies vorwiegend über die sogenannte nicht-homologe Endverknüpfung (engl. non-homologous end joining, NHEJ) und die Homologie-gerichtete Reparatur (engl. homology-directed repair, HDR). Solche Reparaturmechanismen sind wichtig für Zellen, um die Integrität ihres Genoms zu erhalten.
Bei der NHEJ werden mit einer bestimmten Frequenz sogenannte Indel-Mutationen ausgelöst. Dabei werden wenige Basenpaare an die Stelle des Doppelstrangbruchs eingefügt (Insertion) und/oder gelöscht (Deletion). Dies kann zu Punkt- oder Frameshift-Mutationen im betroffenen Gen führen, was den Locus zerstört.
Die HDR benötigt zur Reparatur ein homologes DNA-Stück, dass im Bereich des Doppelstrangbruchs eingefügt wird. Diese Eigenschaft kann genutzt werden, um gewünschte DNA-Abschnitte an den Zielort einzufügen, indem ein homologer Donor-Strang in die Zelle eingebracht wird, der den entsprechenden Abschnitt enthält.
Spezifität
Das CRISPR/Cas-System hat den besonderen Vorteil, dass es auf der Hybridisierung zwischen RNA und DNA beruht. Andere gentechnische Methoden, die Nukleasen zur DNA-Modifikation nutzen, basieren auf der direkten Interaktion zwischen Protein und DNA. Die Spezifität der RNA-DNA-Hybridisierung ist grundsätzlich höher, wodurch das CRISPR/Cas-System die Zielsequenz mit einer sehr niedrigen Fehlerrate erkennt und schneidet.
Grundsätzlich besteht aber bei Anwendung des CRISPR/Cas-Systems die Gefahr, dass sogenannte Off-target-Mutationen entstehen. So werden Veränderungen im Genom bezeichnet, die durch Cas9 außerhalb der eigentlich Zielsequenz hervorgerufen werden.
Anwendung
Das CRISPR/Cas-System gilt als vielversprechende Technologie zur Therapie genetisch bedingter Erkrankungen (Gentherapie). In intestinalen Stammzellen von Mäusen ist es beispielsweise gelungen, die Mutation im CFTR-Gen, die übersächlich für die Mukoviszidose ist, über CRISPR/Cas zu korrigieren.
Quellen
- Sander et al., CRISPR-Cas systems for genome editing, regulation and targeting, Nat Biotechnol; 2014
- Ma et al., Genome modification by CRISPR/Cas9, FEBS J; 2014
- Jinek et al., A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity, Science; 2012
- Schwank et al., Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients, Cell Stem Cell; 2013