Cotranslationaler Proteintransport
Synonym: cotranslationale Proteintranslokation
Englisch: cotranslational protein translocation
Definition
Der cotranslationale Proteintransport ist ein Mechanismus des zellulären Proteintransports, bei dem die Synthese und der Transport miteinander gekoppelt sind. Proteine werden dabei während ihrer Translation in die Membran bzw. das Lumen des ERs transferiert.
Bedeutung
Über den cotranslationalen Proteintransport werden folgende Proteine einer Zelle gebildet: integrale Membranproteine und luminalen Proteine von Organellen des Endomembransystems (ER, Golgi-Apparat, Endosomen, Lysosomen) sowie alle Proteine, die von der Zelle sezerniert werden. Ein großer Teil dieser Proteine wird also nach der Synthese vom ER aus über vesikulären Transport weiter zu anderen Organellen verteilt.
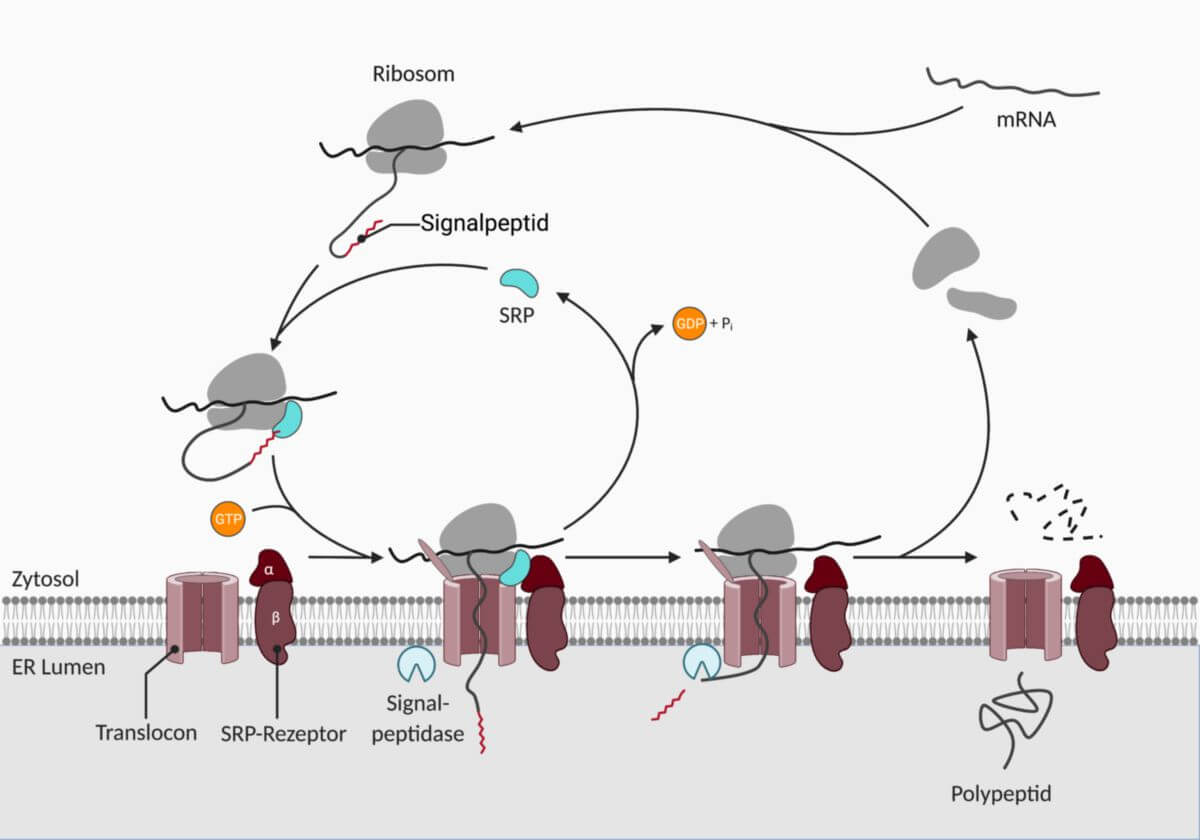
Mechanismus
Proteinerkennung
Proteine, die contranslational transportiert werden, enthalten eine bestimmte Signalsequenz. Diese liegt typischerweise am N-Terminus und enthält 6-12 hydrophobe Aminosäuren. Während der Translation verlässt diese Sequenz das Ribosom als erstes und wird vom Signalerkennungspartikel (SRP) erkannt. Dieses Ribonucleoprotein wiederum bindet den SRP-Rezeptor, der in der ER-Membran liegt. Die Interaktion führt dazu, dass das gesamte Ribosom zum ERs gezogen wird und sich an ein sogenanntes Translocon in der Membran anlagert. Die Bindung von SRP an das Ribosom führt dazu, dass die Translation vorübergehend stoppt.
Transport über die Membran
Das Translocon ist ein Komplex aus mehreren Transmembranproteinen, der einen Kanal in der Membran des ERs bildet. SRP und SRP-Rezeptor binden beide GTP. Sobald das Ribosom auf einem Translocon sitzt, wird das Nukleotid hydrolysiert, wodurch die Bindung an das Ribosom gelöst und die wachsende Polypeptidkette in den Kanal des Translocons geschoben wird. Die Translation wird dann fortgesetzt, bei löslichen Proteinen so lange, bis sich die ganze Kette im Lumen befindet.
Insertion von Membranproteinen
Transmembranproteine besitzen neben der N-terminalen Sequenz für die initiale Erkennung weitere Signalsequenzen innerhalb der Polypeptidkette. Diese Stopp-Sequenzen führen dazu, dass die Translation unterbrochen wird. Das Vorhandensein einer internen Sequenz führt dazu, dass das gesamte Protein lateral vom Translocon in die Membran abgegeben wird. Hieraus resultiert ein Typ-I-Transmembranprotein mit N-terminaler Domäne im Lumen, C-terminaler Domäne im Zytosol und einer Transmembrandomäne (TMD).
Multi-pass-Proteine mit mehreren TMDs besitzen entsprechend viele Stopp-Sequenzen, die allerdings jeweils von einer weiteren Start-Sequenz gefolgt sind. Die Translation stoppt bei der ersten Stopp-Sequenz, die lateral entlassen wird. Die folgende Start-Sequenz initiiert die Fortführung der Translation bis zur nächsten Stopp-Sequenz. Dieser Prozess wiederholt sich so lange, bis alle TMDs in die Membran eingebaut sind.
Proteinfaltung und Modifikation
Das Lumen des ERs enthält verschiedene Chaperone, die die korrekte Faltung der neu synthetisierten Proteine gewährleisten. Hier werden Proteine außerdem durch N-Glykosylierung posttranslational modifiziert.