Basen-Exzisionsreparatur
Englisch: base excision repair (BER)
Definition
Die Basen-Exzisionsreparatur, kurz BER, ist ein DNA-Reparaturmechanismus, der beschädigte DNA-Basen erkennt und ersetzt.
Basenmodifikation
| Basenmodifikation | Beispiel | An Reparatur beteiligte Glykosylase[1] |
|---|---|---|
| Oxidation | Guanin -> 8-Oxoguanin | 8-OxoG DNA glycosylase 1 (OGG1) |
| Thymin -> 5-Hydroxymethyluracil | DNA N-glycosylase activity (HMUDG) | |
| Alkylierung | Guanin -> 7-Methylguanosin | Methylpurine glycosylase (MPG) |
| Adenin -> 3-Methyladenin | Human Alkyladenine DNA Glycosylase (hAAG) | |
| Desaminierung | Cytosin -> Uracil | Uracil-N-Glycosylase (UNG) |
| Guanin -> Xanthin | Human Alkyladenine DNA Glycosylase (hAAG) |
Basenverluste (Depurinierung und Depyrimidinierung), die durch spontane Hydrolyse entstehen können, werden ebenfalls durch die Prozesse der BER verarbeitet, benötigen aber keine Glykosylase.
Mechanismus
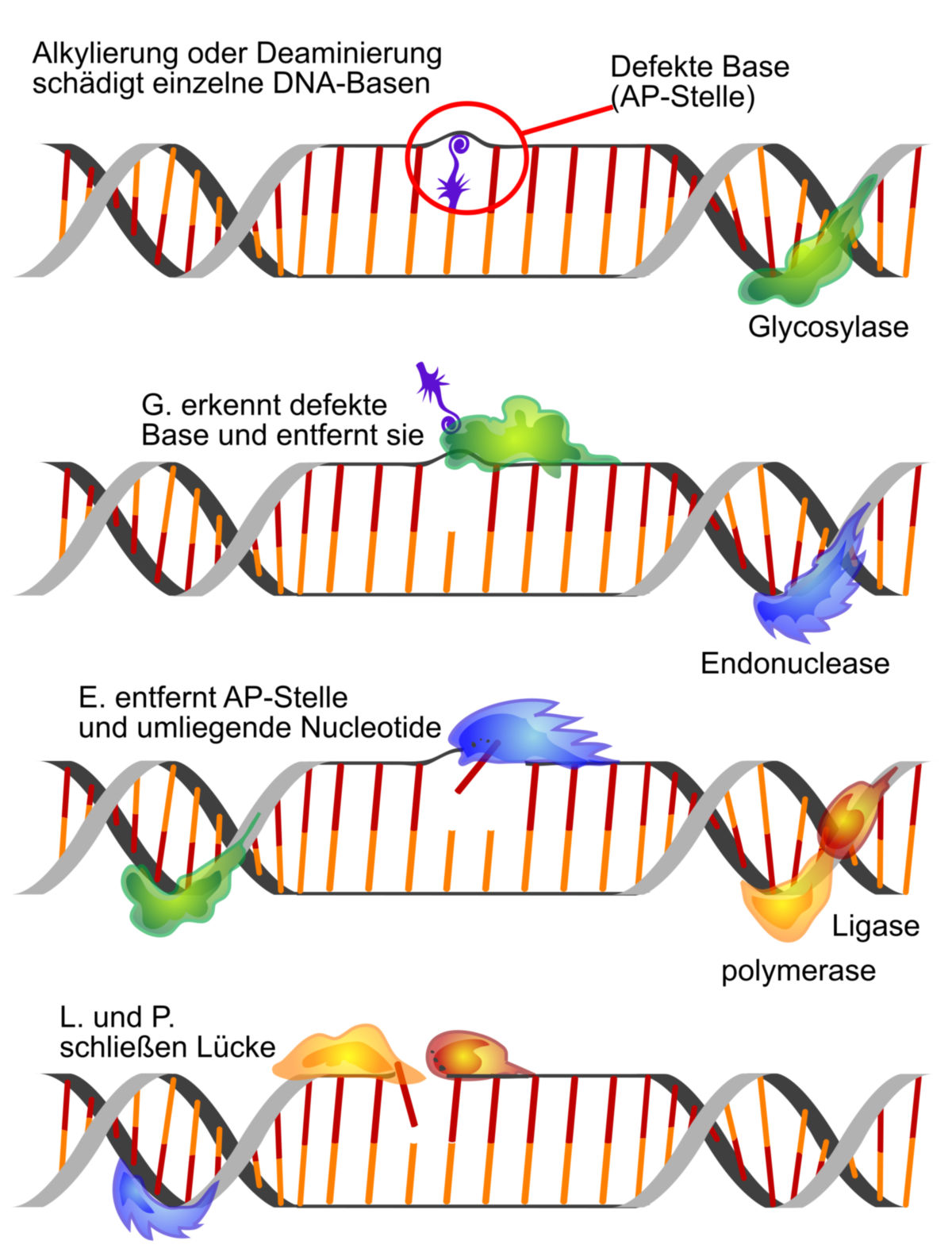
- Eine DNA-Glykosylase trifft auf eine modifizierte Base und entfernt sie. Diese Stellen werden als Apurin- oder Apyrimidin-Stelle (Englisch: "AP-Site") bezeichnet.
- Die AP-Endonuklease 1 (APE1), die Nukleotide ohne Base erkennt, schneidet das Zuckerphosphatrückgrat zusammen mit einer Phosphodiesterase.
- Diese Lücke wird durch DNA-Polymerase β und DNA-Ligase III verschlossen.
- Gelegentlich kommt es auch zur Entfernung von größeren Stücken (Englisch: "Long-Patches") von 2-12 Nukleotiden um die AP-Site herum. Dazu wird außerdem die DNA-Polymerase δ/DNA-Polymerase ε und DNA-Ligase I an die AP-Site rekrutiert.
Der Schlüssel zur Effizienz der BER beruht auf der Vielzahl von verschiedenen Glykosylasen. Diese prüfen einzelne Basen, indem sie diese herausschwenken (Englisch: "flipping-out") und durch Bindung eine Beschädigung feststellen. Der exakte Mechanismus, wie eine einzelne Modifikation innerhalb des Genoms identifiziert wird, ist aber noch nicht endgültig geklärt. Glykosylasen binden meist bevorzugt an eine spezifische Basenmodifikation, können aber auch andere Basenschäden erkennen. In Säugetieren konnten bisher 11 verschiedene Glykosylasen identifiziert werden.[2][3]
Biologischer Hintergrund
Jede menschliche Zelle wird täglich mit tausenden von Basenmodifikationen konfrontiert. Beispielsweise wurden über 20 verschiedene Oxidationsprodukte nachgewiesen.[4] Die häufigste Modifikation, 8-Oxoguanin kann bis zu 1.500 mal pro Tag pro Zelle auftreten. Unreparierte Schäden können zu Transversionen führen.[5]
siehe auch: DNA-Schaden
Quellen
- ↑ Krokan, H. E., Nilsen, H., Skorpen, F., Otterlei, M. & Slupphaug, G. Base excision repair of DNA in mammalian cells. FEBS Letters 476, 73-77, doi:https://doi.org/10.1016/S0014-5793(00)01674-4 (2000).
- ↑ Anthony J.F. Griffiths: Introduction to Genetic Analysis. W.H. Freeman and Company, 2005, Seite 468-463.
- ↑ Jacobs, Angelika L., and Primo Schär. “DNA Glycosylases: In DNA Repair and beyond.” Chromosoma 121.1 (2012): 1–20. PMC. Web. 24 Jan. 2018.
- ↑ Tudek, Barbara et al. “Involvement of Oxidatively Damaged DNA and Repair in Cancer Development and Aging.” American Journal of Translational Research 2.3 (2010): 254–284. Print.
- ↑ Bruce Alberts: Molekularbiologie der Zelle. Wiley-VCH Verlag, 2017, S. 298-301