Ortsspezifische Rekombination
Synonyme: Sequenzspezifische Recombination, Konservative sequenzspezifische Recombination Englisch: Site-specific recombination, conservative site-specific recombination (CSSR)
Definition
Die ortspezifische Rekombination ist ein DNA-Umlagerungsprozess, welcher durch Rekombinasen katalysiert wird. Im Unterschied zur transpositionellen und homologen Rekombination findet sie nur zwischen zwei spezifischen, durch Erkennungssequenzen markierten DNA-Abschnitten statt.
Hintergrund
Der Rekombinationsprozess wird auch als "konservativ" bezeichnet, da es zu keinen Sequenzverdoppelungen oder Verlusten kommt.[1]
Katalysierte Reaktionen
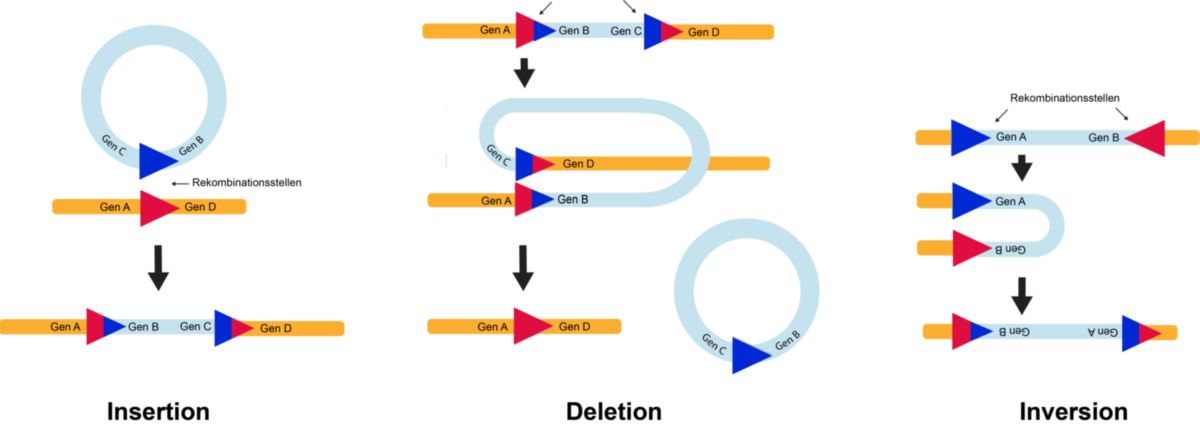
Die beteiligten Rekombinasen können drei mögliche Reaktionen durchführen, welche von der Orientierung der Rekombinationsstellen abhängig sind.
- Insertion: Die Rekombinationsstellen liegen auf zwei verschiedenen DNA-Molekülen und führen zur Integration der Sequenz
- Deletion: Der DNA-Abschnitt zwischen zwei Rekombinationsstellen gleicher Orientierung, welche sich beide auf einem Molekül befinden, wird entfernt und zyklisiert.
- Inversion: Der DNA-Abschnitt zwischen zwei Rekombinationsstellen gegensätzlicher Orientierung wird invertiert.
Ortspezifische Rekombinasen
Die Rekombinasen lassen sich in zwei große, nicht verwandte Gruppen einteilen. Je nach Aminosäurerest im katalytischen Zentrum handelt es sich um Serin- oder Tyrosinrekombinasen. Beide Gruppen bilden in analoger Weise ein DNA/Rekombinasen-Zwischenprodukt, jedoch führen Serinrekombinasen zu Doppelstrangbrüchen, indem alle vier DNA-Stränge gleichzeitig gespalten und wieder zusammengefügt werden. Bei Tyrosinrekombinasen werden je nur zwei Stränge gleichzeitig gespalten und nacheinander zusammengefügt. Für einige Rekombinationssysteme ist ausschließlich die Rekombinase für die Reaktion erforderlich. Meist sind jedoch weitere Hilfsproteine beteiligt.[2]
Anwendung in der Biotechnologie
Die Eigenschaften sequenzspezifischer Rekombinasen können in der Zellkultur und in Modellorganismen eingesetzt werden. Ein bekanntes Beispiel stellt das Cre/loxP-System dar, das aus dem P1-Bakteriophagen abgeleitet wurde. Werden dessen Erkennungsstellen (loxP-sites) vor und hinter einem Gen platziert, so wird die Sequenz dazwischen nach Aktivierung der Cre-Rekombinase entfernt. Die Aktivierung der Cre-Rekombinase lässt sich durch Kombination mit entsprechenden Promotern regulieren. Steht das auszuschaltende Gen beispielsweise unter Kontrolle des CD4-Promotors, so findet ein Knockout fast auschließlich in T-Zellen statt.[3]
Mechanismus
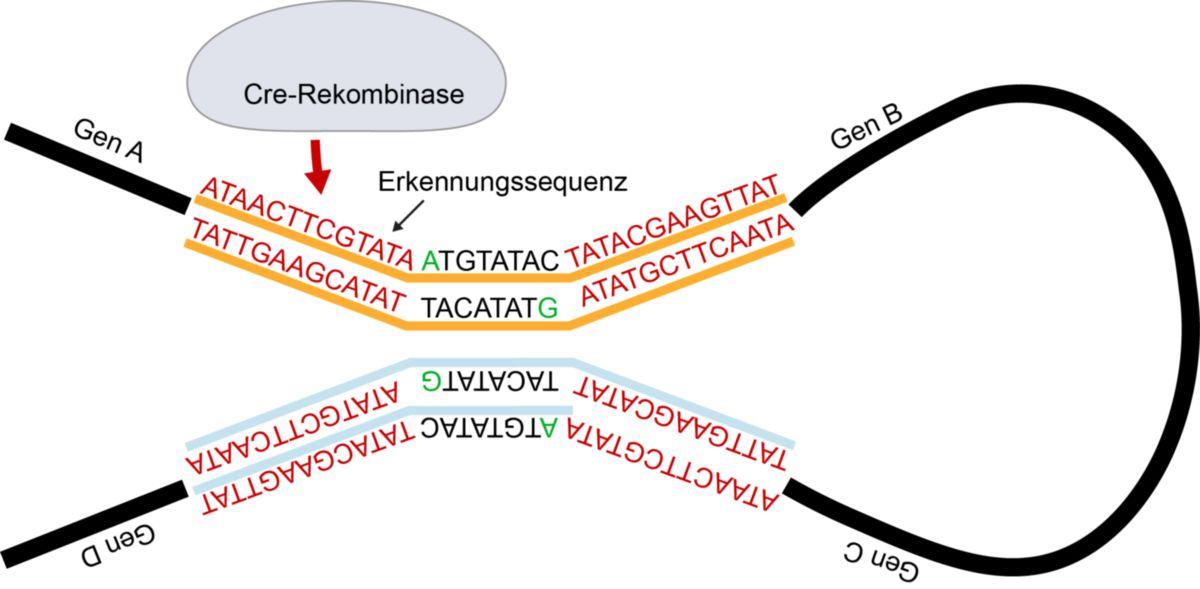
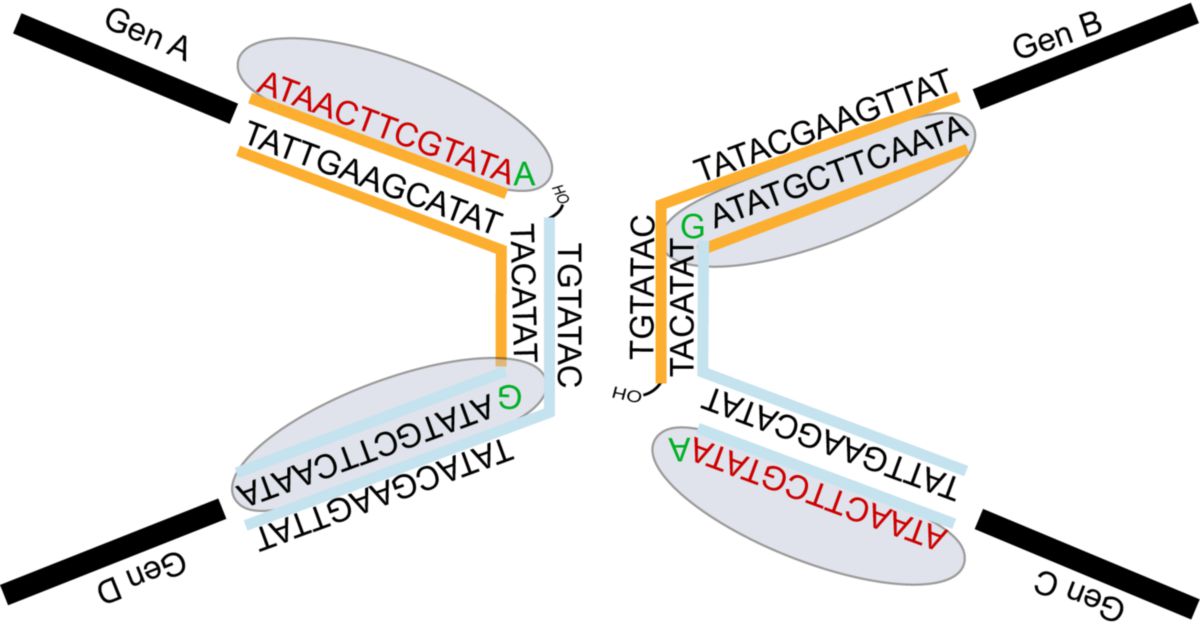
Das folgende Beispiel zeigt den Deletions-Rekombinationsmechanismus der Cre-Rekombinase: Die Rekombinasen binden an die Erkennungssequenzen (hier die loxP-sites)
Ein Tyrosinrest des katalytischen Zentrums greift die Phosphatgruppe des DNA-Rückgrats an. Es bildet sich ein kovalent gebundenes DNA/Rekombinasen Zwischenprodukt.
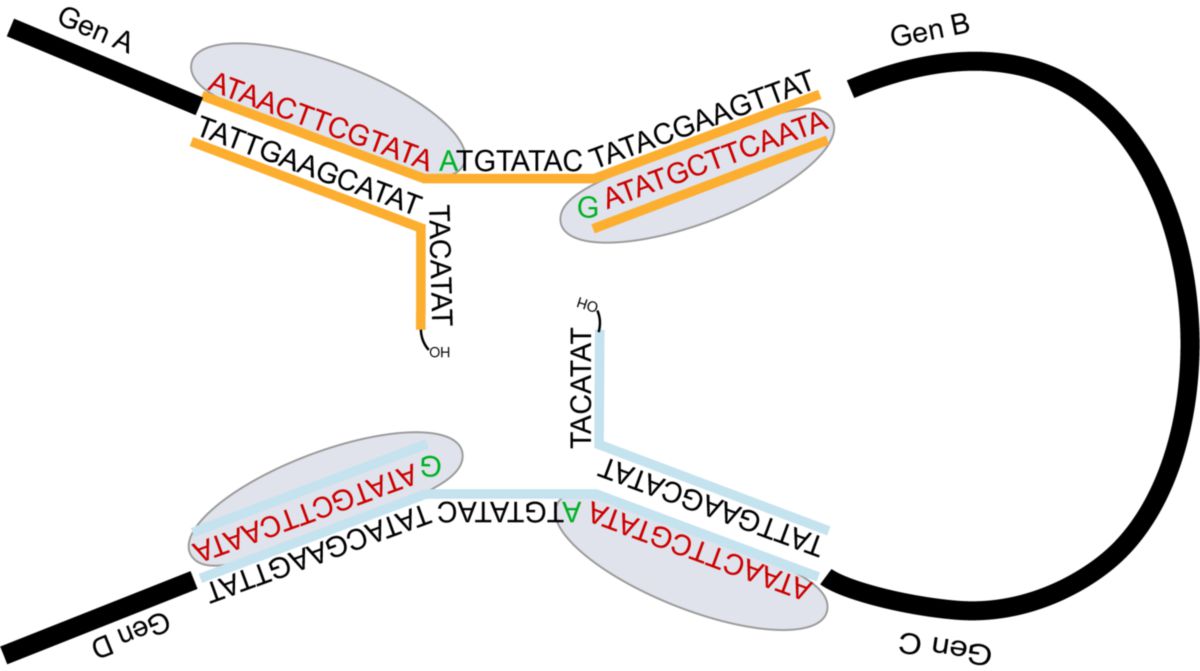
Durch eine Rotation der Rekombinasen-Untereinheiten werden die neuzuverknüpfenden Stränge räumlich koordiniert.
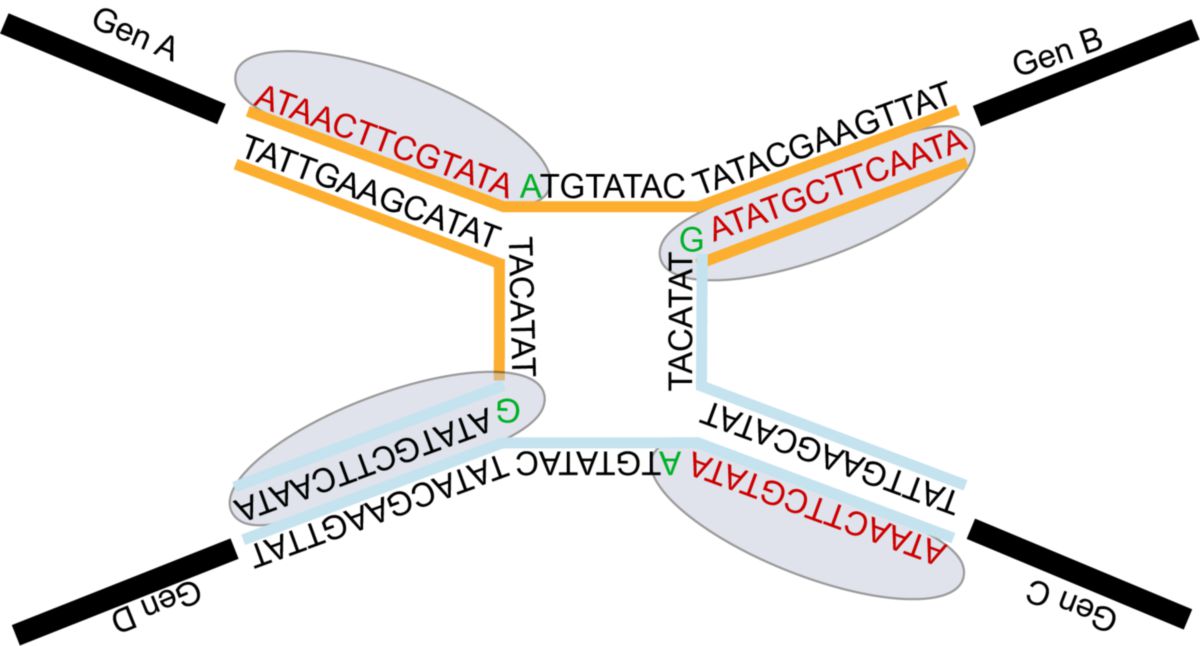
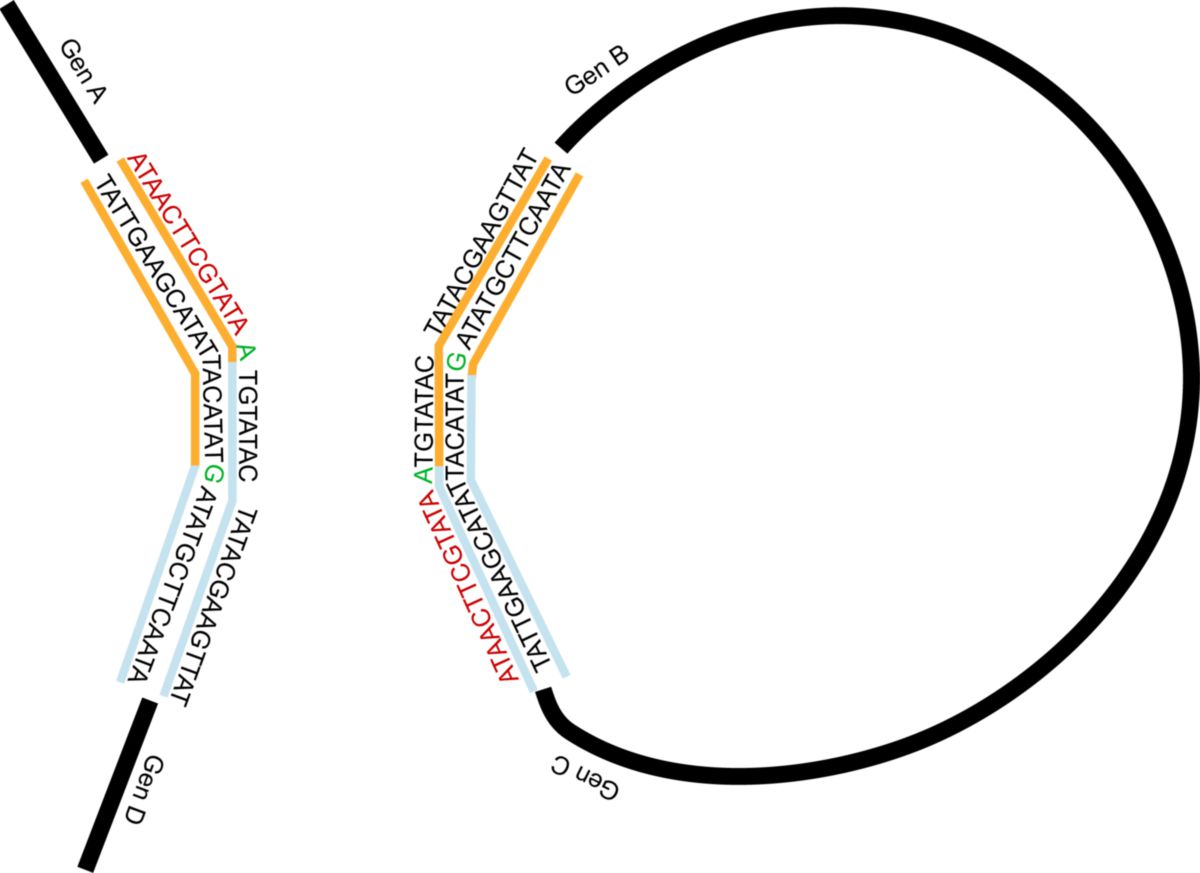
Es erfolgt nun die Neuverknüpfung der Stränge des DNA-Einzelstrang auf den DNA/Rekombinasen-Komplex. Im Fall der Cre-Rekombinase erfolgt dies in zwei Schritten.
Der Abschnitt zwischen den loxP-sites liegt nun als zirkularisiertes Molekül vor.
Quellen
- ↑ James D. Watson: Molekularbiologie. Pearson Studium, 2011, S. 361-365.
- ↑ David P. Clark: Molekulare Biotechnologie. Spektrum Verlag, 2009, S. 419-420.
- ↑ Śledzińska A., Fairbairn L., Buch T. (2014) Manipulation of T Cell Function and Conditional Gene Targeting in T Cells. In: Waisman A., Becher B. (eds) T-Helper Cells. Methods in Molecular Biology (Methods and Protocols), vol 1193. Humana Press, New York, NY