Fehlpaarungsreparatur
Synonyme: Basenmismatch-Reparatur, Mismatch-Reparatur
Englisch: mismatch-repair, MMR
Definition
Die Fehlpaarungsreparatur, kurz MMR, ist ein Mechanismus der DNA-Reparatur, bei dem Fehlpaarungen durch die Replikation erkannt und korrigiert werden.
Hintergrund
Die humane DNA-Polymerase ε, die zusammen mit DNA-Polymerase δ und α für die Replikation benötigt wird, zeigt bereits eine sehr niedrige Fehlerrate. Durch die zusätzliche, als Proofreading bezeichnete 3'-5'-Exonuklease-Aktivität, wird nur jedes 107te Nukleotid falsch gepaart. Die Identifikation der verbleibenden Fehlpaarungen ist aber kritisch für jede Zelle. Mutationen in Proteinen des MMR-Mechanismus sind mit einer Vielzahl von Erkrankungen und einer deutlich erhöhten Krebsdisposition assoziiert.[1]
Die Proteine der MMR sind auch in zahlreiche weitere Signalwege bei DNA-Schäden involviert, z.B. bei der Beseitigung von DNA Modifikationen wie 8-Oxoguanin oder O-6-Methylguanin.
Mechanismus
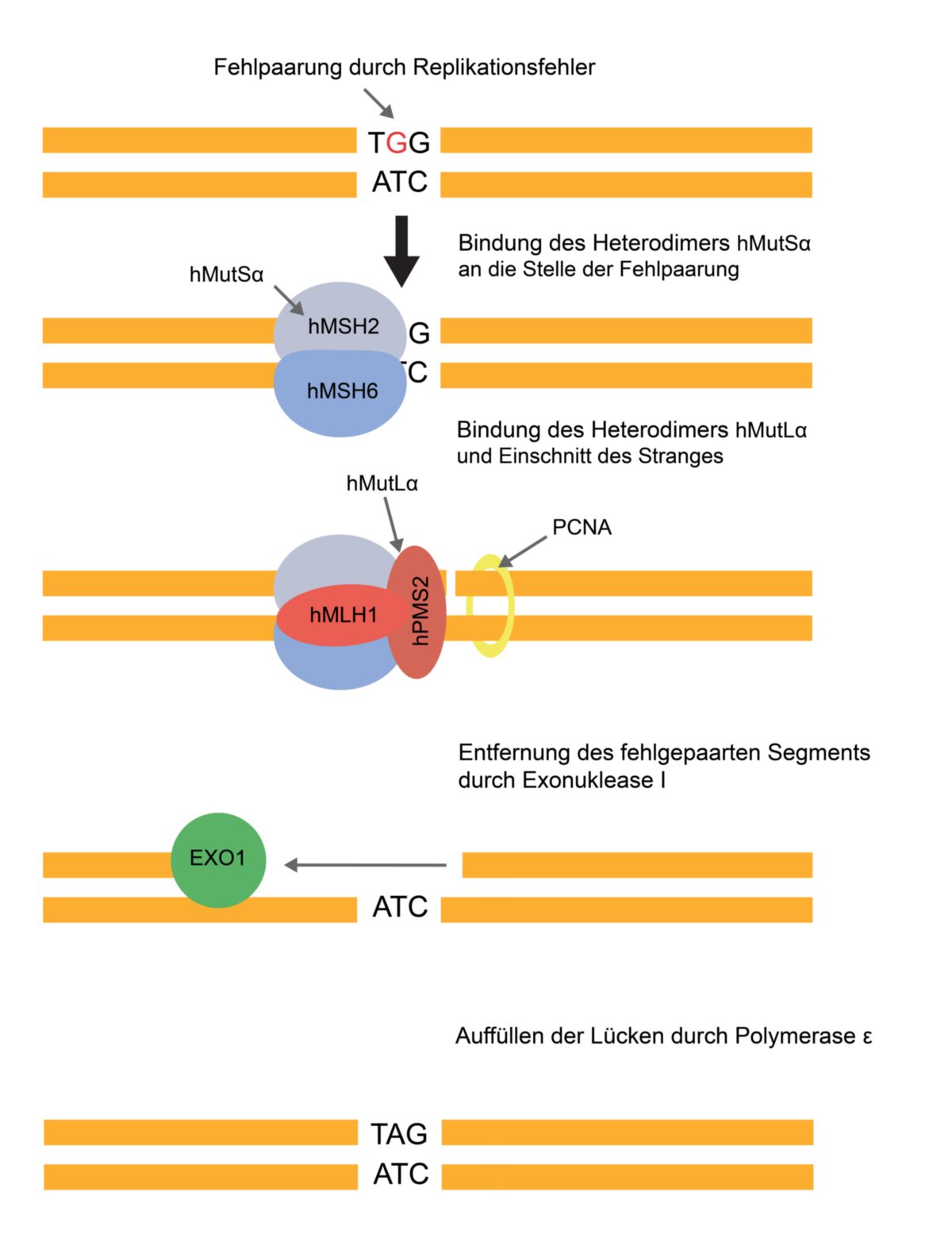
- Das Heterodimer hMutSα, bestehend aus den Untereinheiten hMSH2 und hMSH6, erkennt Basenfehlpaarungen und bindet an diese Stelle.
- hMutLα, bestehend aus den Untereinheiten hMLH1 und hPMS2, erkennt den fehlerhaften (neusynthetisierten) Strang, da dieser kurz nach der Replikation noch einige Stellen besitzt, welche noch nicht durch DNA-Ligasen verschlossen wurden. hMutLα besitzt eine Endonukleasen-Aktivität und schneidet den Strang an der Bindungstelle. Das ringförmige Molekül PCNA ist an der Rekrutierung und richtigen Ausrichtung von hMutLα beteiligt.
- Die rekrutierte Exonuklease EXO1 entfernt das fehlerhafte Segment. Dieser Abschnitt wird durch die DNA-Polymerase δ aufgefüllt und durch DNA Ligase I verschlossen.
Unterschiede zur MMR in E. coli
Der Mechanismus der MMR in Prokaryoten und Eukaryoten ähnelt sich stark und die beteiligten Proteine sind hochkonserviert. Der größte Unterschied besteht in der Identifikation des fehlerhaften Stranges. Bei Escherichia coli funktioniert dies über die Erkennung von DNA-Methylierungen. Kurz nach der Replikation besitzt nur der alte Strang Methylierungen. Das Protein MutH bindet an eine methylierte GATC-Sequenz in der Nähe der Fehlpaarung und beginnt hier den Strang zu schneiden. MutH besitzt im Gegensatz zu MutS und MutL kein Homolog in Eukaryoten.[2]
Klinik
Fehlpaarungsreparaturen führen zur Aktivierung von Tumorsuppressoren wie p53. Defekte der relevanten MMR-Proteine sind darüber hinaus für die Akkumulation von klinisch relevanten Mutationen verantwortlich.
Als bekanntestes Beispiel gilt die Mikrosatelliteninstabilität beim Lynch-Syndrom, die durch eine fehlerhafte MMR verursacht wird. Ein weiteres Beispiel ist das CMMRD-Syndrom.[3]
Quellen
- ↑ Henninger, E. E. and Pursell, Z. F. (2014), DNA polymerase ε and its roles in genome stability. IUBMB Life, 66: 339–351. doi:10.1002/iub.1276
- ↑ Marinus, M.G. "DNA Mismatch Repair." EcoSal Plus 5.1 (2012): 10.1128/ecosalplus.7.2.5. PMC. Web. 20 Jan. 2018
- ↑ Karran, P. 2001. Human Mismatch Repair: Defects and Predisposition to Cancer.