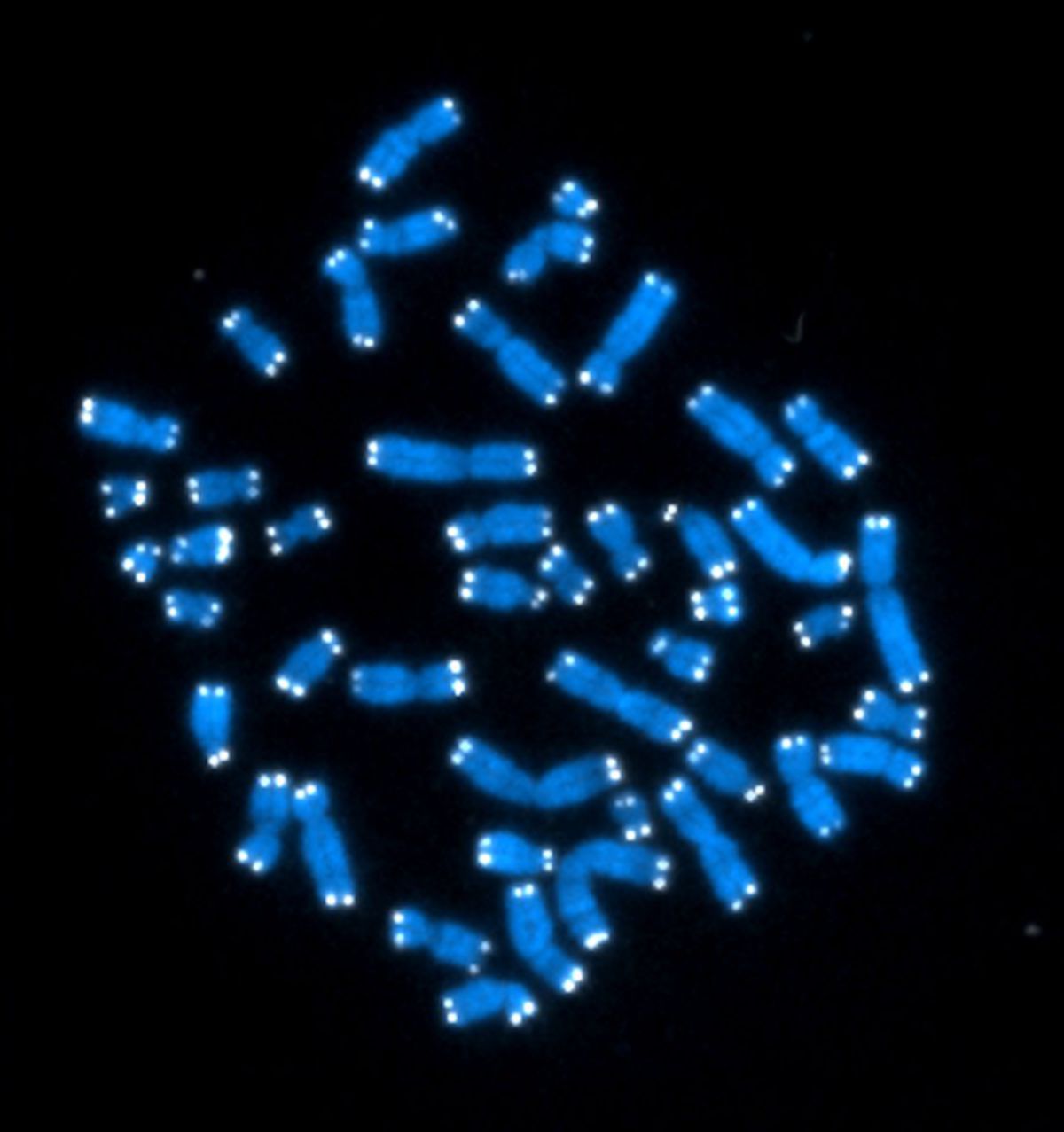
Fluoreszenz-in-situ-Hybridisierung
Abkürzung: FISH
Englisch: fluorescence in situ hybridization
Definition
Bei der Fluoreszenz-in-situ-Hybridisierung, kurz FISH, handelt es sich um ein Verfahren der Zytogenetik, das dem Nachweis von Chromosomenaberrationen dient, welche dem Nachweis durch ein konventionelles Karyogramm entgehen.
Prinzip
Man verwendet Fluoreszenz-markierte DNA-Sonden, die komplementär zu einem gesuchten DNA-Abschnitt sind. Diese Sonden werden zu denaturierter DNA (durch Alkalisierung oder Hitze) hinzugegeben und können dann an den komplementären Abschnitt binden (Hybridisierung). Meist ist die Bindung hochspezifisch für einen bestimmten Chromosomenabschnitt. Die Bindung an den DNA-Abschnitt bzw. dessen Ausbleiben kann mittels Fluoreszenzmikroskop nachgewiesen werden. Das gibt Aufschluss darüber, ob der betreffende Abschnitt vorhanden ist, wo er lokalisiert ist und wie häufig er vorkommt.
Durch die FISH-Technik kann eine Vielzahl von Ziel-DNAs durch unterschiedlich markierte DNA-Sonden in Kombination mit verschiedenen Fluoreszenzfarbstoffen gleichzeitig mit Hilfe einer besonderen Auswertungssoftware analysiert werden, was als Multicolor-FISH (M-Fish) bezeichnet wird. Das ermöglicht auch die simultane, differenzielle Darstellung von allen 22 Autosomen und den 2 Gonosomen (24-Farben-Karyotypisierung) oder auch die Darstellung von Banden eines Chromosomes (Multicolor Banding, MCB).
Markierung von Telomeren auf Chromsomen mittels FISH
Einteilung
Es sind folgende FISH-Methoden möglich:
- Direkter Nachweis: Ein Fluoreszenzfarbstoff (z.B. Dinitrophenol) ist direkt an eine DNA-Sonde gebunden, so dass die mikroskopische Analyse der Ziel-DNA sofort nach der Hybridisierung erfolgen kann.
- Indirekter Nachweis: Die DNA-Sonde ist nicht direkt mit Fluoreszenzfarbstoff markiert, sondern mit einem Stoff wie Biotin oder Digoxigenin, an den erst nach Hybridisierung mit der Ziel-DNA fluoreszierende Stoffe (Avidin oder spezielle Antikörper) binden können. Diese indirekte Methode ist komplizierter, aber sensitiver.
DNA-Sonden
Je nach Fragestellung können verschiedene DNA-Sonden eingesetzt werden. Man unterscheidet primär folgende Typen:
- Chromosomenspezifische Zentromer-DNA-Sonden: Zum Nachweis numerischer Chromosomenveränderungen (mehrere 1000 kbp, beim Interphase-Kern)
- Lokusspezifische DNA-Sonden: Sie markieren chromosomenspezifische Zielsequenzen (5-200 kbp), sprich bestimmte Gene, Chromosomensegmente oder Chromosomenarme und eignen sich zum Nachweis von kleinen Mutationen (Deletionen, Duplikationen, Translokationen) im Metaphase-Kern, aber auch zum Nachweis von numerischen Aberrationen im Interphase-Kern.
- Chromosomenspezifische DNA-Library-Sonden: Erfassung von Translokationen und Insertionen durch spezifische Markierung eines einzelnen Chromosoms ("whole chromosome painting", WCP) mittels vielen kleinen Einzelsonden.
- Vergleichende Genomhybridisierung (comparative genomic hybridization, CGH): Umfassende Analyse von Zugewinn/Verlust an chromosomalem Material, in dem DNA aus Gewebe und Kontroll-DNA um Hybridisierungsstellen in einem Metaphasenchromosom konkurrieren und die Menge an DNA-Material anhand der Stärke des Fluoreszenzsignals geprüft wird.
Beispiel
Pränataldiagnostik bei Verdacht auf ein Down-Syndrom: hier verwendet man DNA-Sonden, die an Chromosom 21 binden können und es sichtbar machen (wenn es 3-fach - nicht wie normal 2-fach - vorliegt, handelt es sich um das Down-Syndrom).