T7-Endonuklease-I-Assay
Synonyme: T7 Assay, Enzyme Mismatch Cleavage, EMC
Englisch: T7 endonuclease I assay
Definition
Der T7-Endonuklease-I-Assay, kurz T7EI, ist eine molekularbiologische Methode zum Nachweis von DNA-Fehlpaarungen (Mismatches). Er wird zur Abschätzung der Effektivität von genommodifizierenden Methoden (Genome Editing) wie CRISPR/Cas9, aber auch zur Mutationsanalyse zahlreicher Organismen eingesetzt.
Hintergrund
Moderne Methoden des Genome Editing können zielgerichtet Mutationen innerhalb eines DNA-Abschnitts einführen. Sowohl Zinkfingernukleasen (ZFN), TALENs als auch CRISPR/Cas9 führen zu Doppelstrangbrüchen, die durch fehleranfällige Reparaturmechanismen (NHEJ) zusammengefügt werden können. Hierbei können Insertionen oder Deletionen auftreten.
Mit dem T7EI-Assay kann schon zu einem frühen Zeitpunkt eines Genome-Editing-Experiments überprüft werden, ob das entwickelte Konstrukt die Zielsequenz effektiv schneidet. Für einen CRISPR/Cas9-Ansatz beispielsweise werden meist pro Zielsequenz mehrere gRNAs designt, da auch geeignete Software die Bindungsfähigkeit nicht zuverlässig vorhersagen kann.[1]
Prinzip
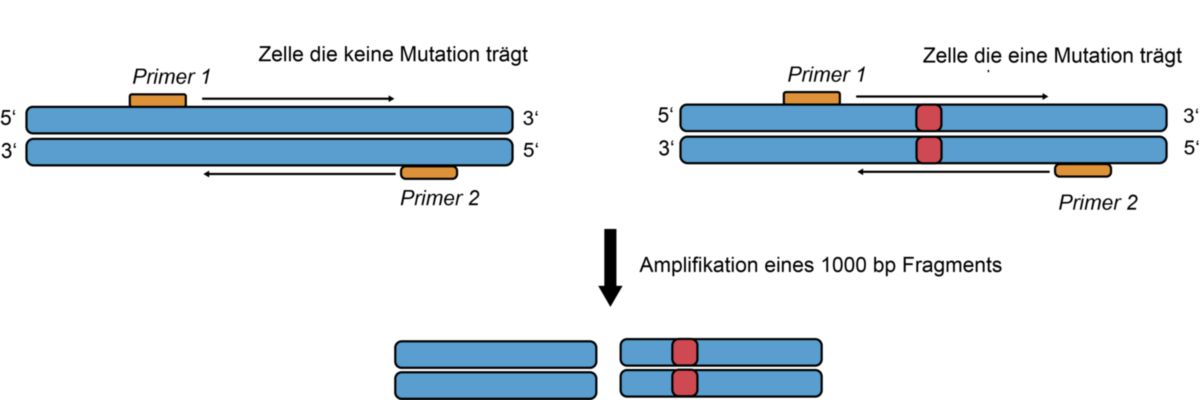
Der T7EI-Assay basiert auf der Fähigkeit der T7-Endonuklease I, auch einzelne Basenfehlpaarungen zu erkennen und an dieser Stelle die DNA zu schneiden. Lagert sich also ein Wildtyp-Einzelstrang an einen mutierten an, so bilden diese einen Heteroduplex, der an der Position des Mismatches Strukturveränderungen aufweist, die von der Endonuklease erkannt werden können. Mittels Gelelektrophorese können die zwei Fragmente nachgewiesen werden.[2]
Durchführung
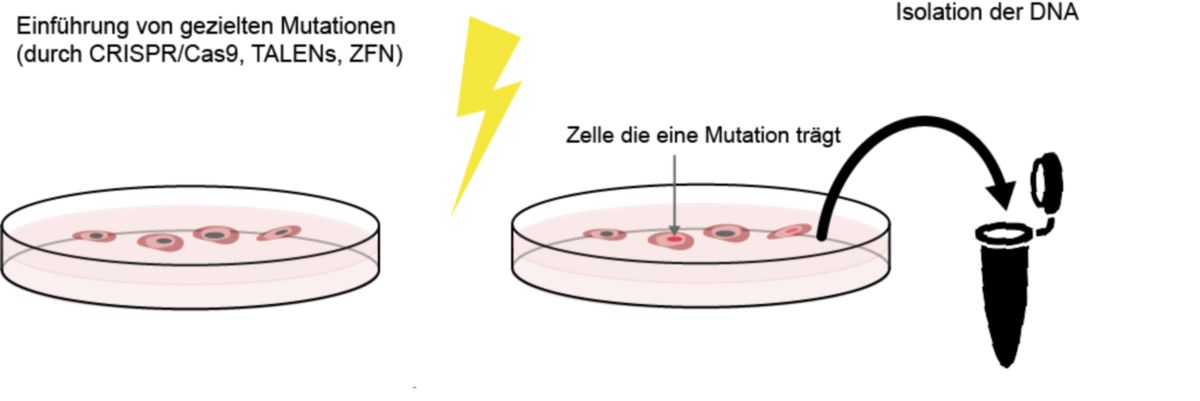
DNA-Isolation:
- Die DNA wird aus den Zielzellen isoliert.
Fragment-Amplifikation:
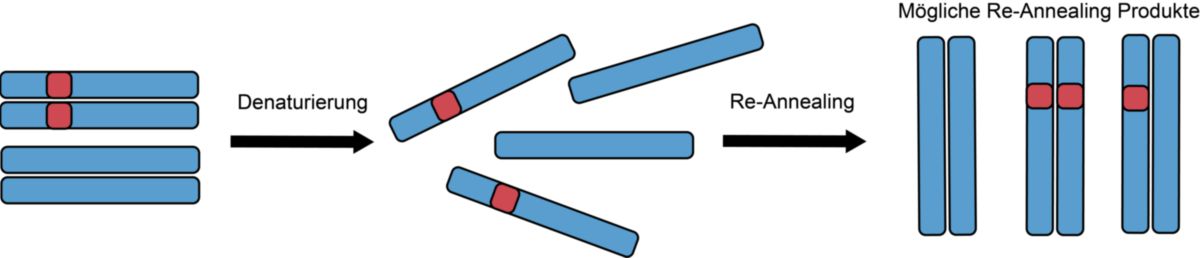
Heteroduplex-Annealing:
- Der Reaktionsansatz wird auf 96 °C erhitzt, gefolgt von einer langsamen, schrittweisen Temperaturabsenkung auf Raumtemperatur. Dadurch werden zuerst die Stränge getrennt, welche sich nachfolgend wieder zufällig anlagern. Es können drei Produkte entstehen:
- ein Wildtyp-Wildtyp-Fragment (Homoduplex),
- ein Wildtyp-Mutant-Fragment (Heteroduplex) und
- ein Mutant-Mutant-Fragment (Homoduplex)
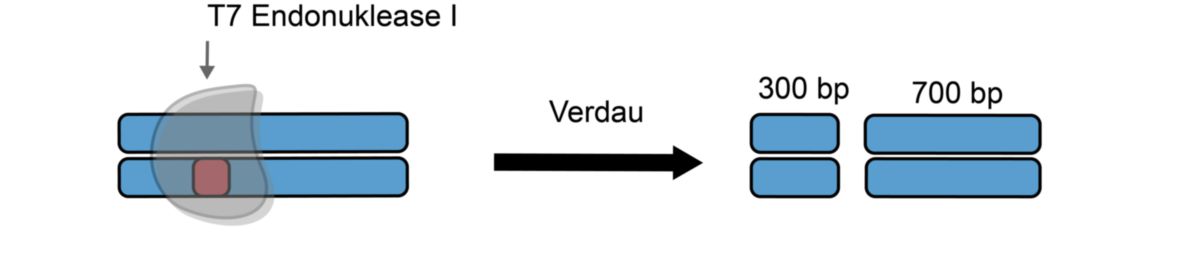
T7-Endonuklease-I-Verdau:
- Die Reaktion wird mit der Endonuklease I inkubiert. Duplexe, die Fehlpaarungen enthalten, werden nun zerschnitten. Die Fragmente können mittels Gelelektrophorese oder Kapillarelektrophorese getrennt und analysiert werden.
Interpretation
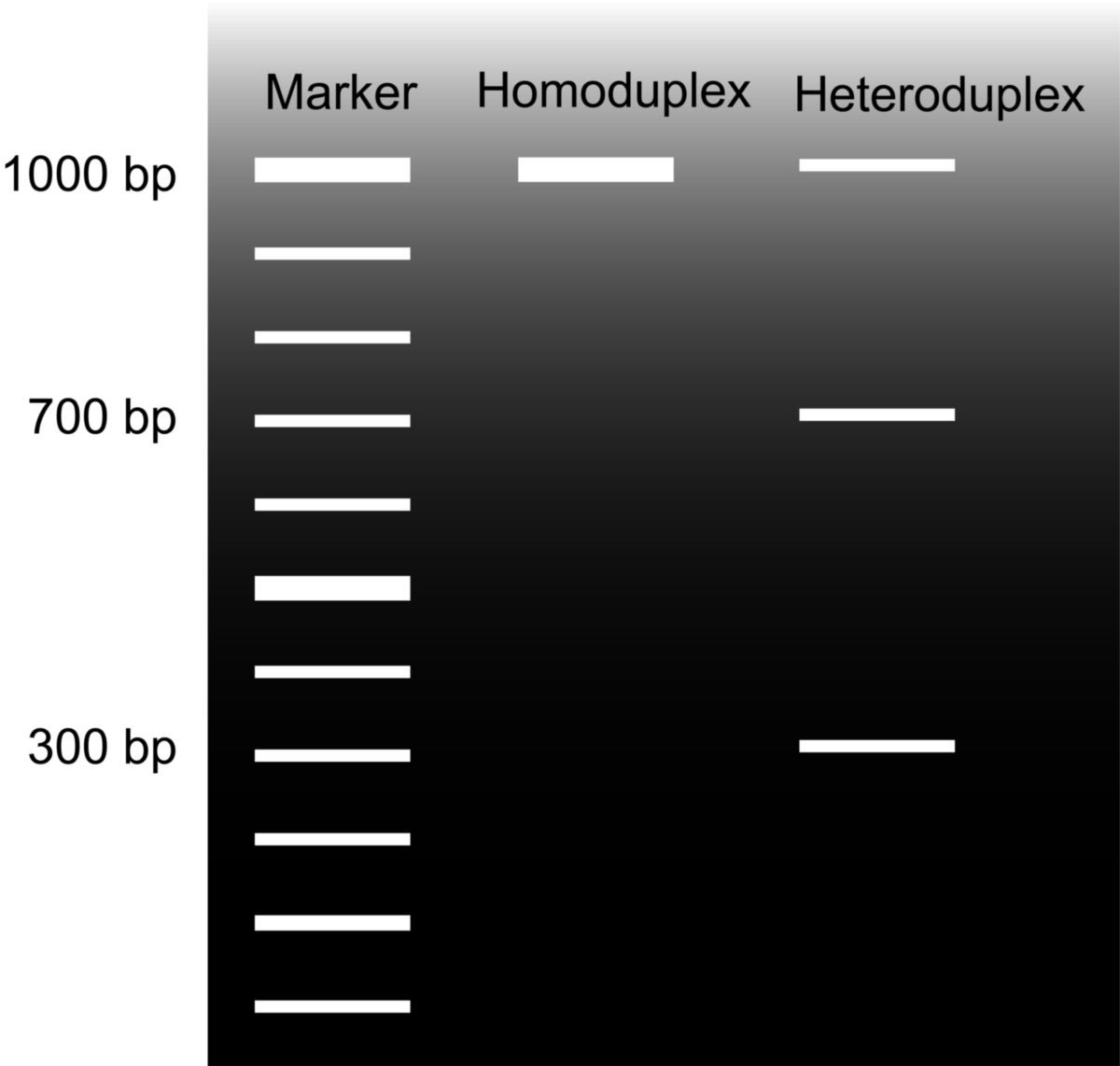
Die folgende Abbildung zeigt eine Auswertung mittels Gelektrophorese. In der ersten Probe ist nur eine Bande zu sehen, es hat also keine Mutation stattgefunden. In der zweiten Probe sind sowohl der ungeschnittene Homoduplex (1.000 bp), das geschnittene längere Fragment (700 bp) und das geschnittene kürzere Fragment (300 bp) zu sehen. Nur das zweite Konstrukt sollte also für weitere Experimente verwendet werden. Eine exakte Quantifizierung der Genom-Targeting-Effizienz kann mittels Kapillarelektrophorese erfolgen.
Quellen
- ↑ Vouillot, Léna, Aurore Thélie, and Nicolas Pollet: Comparison of T7E1 and Surveyor Mismatch Cleavage Assays to Detect Mutations Triggered by Engineered Nucleases. G3: Genes|Genomes|Genetics 5.3 (2015): 407–415. PMC. Web. 6 Oct. 2017.
- ↑ Guschin, D.Y., et. al.: A rapid and general assay for monitoring endogenous gene modification. Methods Mol Biol, 649, 247–256.